|
||||||||||||||||||||||||||||||||||||
|
基于深度学习识别RiPPs前体肽及裂解位点
合成生物学
2022, 3 (6):
1262-1276.
DOI:10.12211/2096-8280.2022-016
得益于基因测序技术的快速发展,基因组测序数据呈现爆炸式增长,核糖体合成和翻译后修饰肽(RiPPs)是近十年逐渐进入人们视野的一大类肽类天然产物。这类化合物在自然界中分布极其广泛,具有丰富的结构多样性和生物活性多样性,是天然药物的重要来源。RiPPs的发现主要依赖低通量生物实验,传统方法精确但成本高昂,随着新型计算机技术的更新迭代,包括antiSMASH、RiPP-PRISM等在内的生物信息学工具能够极大加速RiPPs挖掘进程,但依然无法突破基于同源性方法(例如搜索保守的生物合成酶)的限制——无法有效识别具有不同生物合成机制的新型RiPPs。在这里,本文首次基于自然语言处理预训练模型BERT,提出四种可以完全依赖序列数据识别RiPPs而非基于同源性及基因组上下文信息的深度学习模型,通过对各模型进行验证分析和对比,最终确定在RiPPs识别赛道上表现卓越的最佳模型BERiPPs(bidirectional language model for enhancing the performance of identification of RiPPs precursor peptides)。BERiPPs能够在不考虑基因组背景的情况下以无偏见的方式识别RiPPs前体肽,并可通过条件随机场生成对前导肽裂解位点的预测,为高通量挖掘全新RiPPs提供了思路,并在一定程度下揭示了前体肽和修饰酶间的生物学底层关系。
表1
不同算法在RiPPs前体肽识别任务上的效果对比
正文中引用本图/表的段落
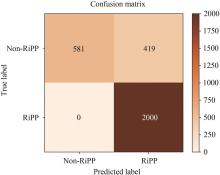
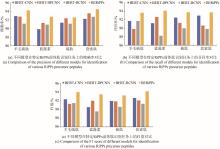
在同一个数据集上,分别采用BERT、BERT-CNN、BERT-DPCNN、BERT-RCNN、BERT-BiLSTM模型进行训练,在对模型超参数多次优化后得到各模型在RiPPs前体肽识别任务上的预测性能测试结果,如表1所示。
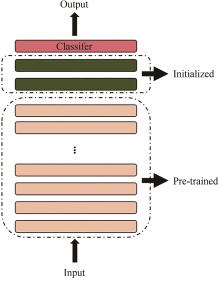
可以看到,直接将预训练模型BERT用于RiPPs前体肽识别也能取得较好的预测效果,这不仅体现了BERT在自然语言处理上的强大,也有理由相信BERT在基于特定领域调整后会得到理想的效果。但令人意外的是,不管是在将BERT顶部1层还是2层参数重新初始化后,模型的预测性能都没有较大的变化,只是在训练中收敛速度略微加快,而基于完全随机初始化后的BERT重新训练,最终结果比预训练后的BERT精确率低了3.2%,基于对比结果不妨可以合理地做出推测,BERT在特殊领域下的较好性能表现不仅源于通过预训练得到的初始化参数相较于随机初始化对特征抽取性能的提升,其强大的学习能力也是关键因素之一。在将BERT作为embedding层,与其他主流模型组合后发现识别效果较BERT原模型有了一定提升,且与RNN模型组合后的预测性能整体上要优于CNN,这也从侧面反映了RNN在处理文本序列上较CNN具有更好的性能表现。其中,由BERT与BiLSTM组合的BERiPPs模型明显优于其他组合模型,其精确率、召回率和F1值分别为93.31%、91.70%、92.50%。
式中,
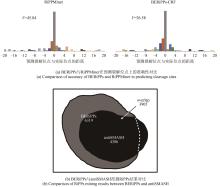
BERiPPs-CRF模型通过对氨基酸序列的标注进行识别,从而间接生成对RiPPs前体肽裂解位点的预测。从实体识别的角度来说,BERiPPs-CRF依然表现了较高的水平,精确率、召回率和F1值分别为90.45%、91.33%和90.88%。但从对RiPPs前体肽裂解位点预测的角度来看,其重点在于能否准确判断标签B(即核心肽起始氨基酸)所在的位置。同样受限于数据集中各类RiPPs样本的数量以及不同RiPPs家族所展现的前体切割规则的差异,在对RiPPs前体肽裂解位点的预测上准确率出现了明显的两极差异。例如对套索肽裂解位点的准确率达到了70%以上,Ⅰ、Ⅱ型羊毛硫肽裂解位点的预测准确率也超过了60%,而小型的RiPPs家族的预测结果则不太理想。如果把与真实裂解位点相差±5个氨基酸的预测也纳入考虑范围之内,那么整体预测的准确率为80.67%。本文将基于机器学习的RiPPMiner用同样的测试数据集进行预测,与BERiPPs-CRF模型对比结果如图8(a)所示。对于没有准确识别真实裂解位点的样本而言,模型预测的位点与实际位点相隔越近,则越有RiPPs研究的借鉴意义,如果模型能够做到把预测与真实位点之间的间隔控制在±5甚至±3、±1个氨基酸以内,其模型的价值也将按倍数增加。因此,为了更直观地展现预测位点与实际位点的偏差程度,本文根据统计学规则引入一个新的评估指标偏位度
本文的其它图/表
|