|
||
|
基于深度学习识别RiPPs前体肽及裂解位点
合成生物学
2022, 3 (6):
1262-1276.
DOI:10.12211/2096-8280.2022-016
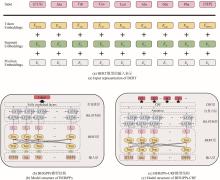
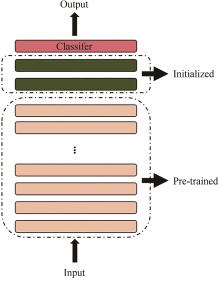
得益于基因测序技术的快速发展,基因组测序数据呈现爆炸式增长,核糖体合成和翻译后修饰肽(RiPPs)是近十年逐渐进入人们视野的一大类肽类天然产物。这类化合物在自然界中分布极其广泛,具有丰富的结构多样性和生物活性多样性,是天然药物的重要来源。RiPPs的发现主要依赖低通量生物实验,传统方法精确但成本高昂,随着新型计算机技术的更新迭代,包括antiSMASH、RiPP-PRISM等在内的生物信息学工具能够极大加速RiPPs挖掘进程,但依然无法突破基于同源性方法(例如搜索保守的生物合成酶)的限制——无法有效识别具有不同生物合成机制的新型RiPPs。在这里,本文首次基于自然语言处理预训练模型BERT,提出四种可以完全依赖序列数据识别RiPPs而非基于同源性及基因组上下文信息的深度学习模型,通过对各模型进行验证分析和对比,最终确定在RiPPs识别赛道上表现卓越的最佳模型BERiPPs(bidirectional language model for enhancing the performance of identification of RiPPs precursor peptides)。BERiPPs能够在不考虑基因组背景的情况下以无偏见的方式识别RiPPs前体肽,并可通过条件随机场生成对前导肽裂解位点的预测,为高通量挖掘全新RiPPs提供了思路,并在一定程度下揭示了前体肽和修饰酶间的生物学底层关系。  View image in article
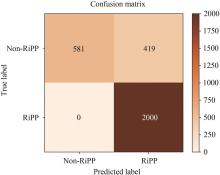
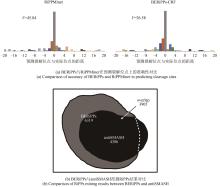
图7
不同训练方式下的BERiPPs和DeepRiPP在预测RiPPs类别上的结果对比
(因测试集中各类RiPPs样本数量不同,故将混淆矩阵中的数值进行归一化处理,再根据四舍五入原则精确到小数点后一位)
正文中引用本图/表的段落
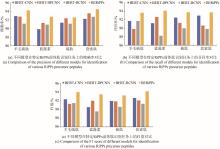
像antiSMASH这样的基因簇分析工具可以根据BGCs中修饰酶来预测RiPPs的类别,例如合成羊毛硫肽的脱水环化酶以及套索肽的天冬酰胺合成酶,这样以修饰酶为中心来搜索可能的相邻前体短肽的策略可以使antiSMASH快速分析BGCs中是否存在RiPPs以及RiPPs的类别,但这种策略只能局限于已知的生物合成途径范围内,而BERiPPs仅通过RiPPs前体的氨基酸序列训练的深度学习模型来生成对RiPPs类别的预测,在对RiPPs精准识别的基础上进一步拓展。 BERiPPs训练的结果符合预期,在例如各型羊毛硫肽、套索肽及硫肽等大型RiPPs类的识别上准确率较高,但受限于小类RiPPs数据集十分有限,对林那肽类、赛克肽类等的预测精度较低。对此,本文采用k折交叉验证以及留出法(hold-out method)[32]结合的方式,将原始数据集按照9∶1的比例分为A、B数据集。之后将数据集A等分为9部分,每次采用不同的部分作为验证集,其余数据均作为训练集以此训练并验证模型,重复9次后再将作为hold-out set的数据集B分别用于测试模型BERiPPs。这样可保证数据集除hold-out set以外的所有数据都能参与模型的训练,使模型对于数据的划分不那么敏感。在目前采用深度学习挖掘RiPPs的主流工具中,NeuRiPP将重点更多地聚焦于在大量非RiPPs负例样本中准确地识别RiPPs[17],为了更直观地评估BERiPPs在RiPPs类别预测上的性能,本研究选取了DeepRiPP在同样的测试集上的预测结果作为对比,如图7所示训练方式的优化在一定程度上提高了BERiPPs的预测性能和泛化能力。同时,DeepRiPP也展现出了强大的RiPP识别能力,与BERiPPs在对不同类别的识别性能上各有优劣,但比较明显的是在对林那肽类的预测能力上明显低于优化后的BERiPPs。
本文的其它图/表
|