Synthetic methylotrophy: engineering the production of biofuels and chemicals based on the biology of aerobic methanol utilization
2
2015
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
... [1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Hydrogenation of CO2 to methanol over CuCeTiO x catalysts
1
2017
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Research progress on the indirect hydrogenation of carbon dioxide to methanol
1
2016
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Renewable methanol and formate as microbial feedstocks
1
2020
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Synthetic methylotrophy: strategies to assimilate methanol for growth and chemicals production
1
2019
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Guidance for engineering of synthetic methylotrophy based on methanol metabolism in methylotrophy
3
2017
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
... [6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
... 试剂:NBT(硝基四氮唑蓝)、PMS(酚嗪硫酸甲酯)、溶菌酶、MTT(噻唑蓝)、PES(酚嗪硫酸乙酯)、NAD等购自北京鼎国昌盛生物技术公司.NCD由本实验室合成[6]. ...
A new alcohol dehydrogense, reactive towards methanol, from Bacillus stearothermophilus
3
1988
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
... [7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
... (2)构建基于甲醇的新型氧化还原催化体系.甲醇可分3步逐级生物氧化为CO2,每步反应均有相应辅酶依赖型氧化还原酶从而产生计量的还原型辅酶.本研究得到NCD偏好型MDH将甲醇氧化为甲醛,而此前的研究已获得NCD偏好型甲酸脱氢酶[29,46].未来如果将甲醛脱氢酶的辅酶偏好性也予以改造并获得NCD偏好型突变体,即可构建以甲醇为还原性底物的NCDH供给体系,用于选择性为特定代谢反应或途径提供还原力.本课题组认为,这种基于甲醇的NCDH供给体系,与此前构建的基于亚磷酸[7]和甲酸[8]的体系相比,更加绿色并具有更好的经济性. ...
Engineering the biological conversion of methanol to specialty chemicals in Escherichia coli
3
2017
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
... 构建文库及蛋白质表达及纯化时所用宿主为大肠杆菌BL21(DE3).密码子优化的BsMDH编码基因[8]由Synbio Technologies合成,并已克隆在表达质粒pTrc99K-MDH[32]. ...
... (2)构建基于甲醇的新型氧化还原催化体系.甲醇可分3步逐级生物氧化为CO2,每步反应均有相应辅酶依赖型氧化还原酶从而产生计量的还原型辅酶.本研究得到NCD偏好型MDH将甲醇氧化为甲醛,而此前的研究已获得NCD偏好型甲酸脱氢酶[29,46].未来如果将甲醛脱氢酶的辅酶偏好性也予以改造并获得NCD偏好型突变体,即可构建以甲醇为还原性底物的NCDH供给体系,用于选择性为特定代谢反应或途径提供还原力.本课题组认为,这种基于甲醇的NCDH供给体系,与此前构建的基于亚磷酸[7]和甲酸[8]的体系相比,更加绿色并具有更好的经济性. ...
Characterization and evolution of an activator-independent methanol dehydrogenase from Cupriavidus necator N-1
1
2016
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
一碳代谢关键酶——甲醇脱氢酶的研究进展与展望
1
2021
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Methanol dehydrogenase, a key enzyme of one-carbon metabolism: a review
1
2021
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Engineering Corynebacterium glutamicum for methanol-dependent growth and glutamate production
1
2018
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Growth of E. coli on formate and methanol via the reductive glycine pathway
1
2020
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Complete oxidation of methanol in biobattery devices using a hydrogel created from three modified dehydrogenases
1
2013
... 甲醇是大宗有机一碳化合物,全球年产量达6000万吨,有望成为未来生物炼制产业的重要原料[1-2].煤、天然气、页岩气和CO2等通过化学法可规模化制备甲醇,而在废物处理和垃圾填埋场中,厌氧消化产生的甲烷和合成气,也可进一步转化为甲醇[3].通过改造甲基营养型微生物或重建甲基利用途径,构建了一系列以甲醇为原料的微生物细胞工厂[4-5].甲醇氧化酶将甲醇氧化为甲醛为代谢提供碳源,同时产生过氧化氢,后者需要消耗额外能量予以清除[6].烟酰胺腺嘌呤二核苷酸(NAD,图1)依赖型甲醇脱氢酶(MDH)也可氧化甲醇合成甲醛,同时生成化学计量的NADH可作为代谢的还原力[7].来源于嗜热脂肪芽孢杆菌Bacillus stearothermophilus DSM2334的NAD依赖型MDH(BsMDH,UniProt:P42327.1)对甲醇亲和力较高[8],Km为20 mmol/L[7].目前文献中研究较多的NAD依赖型MDH包括BsMDH和Bacillus methanolicus(BmMDH)[6],而BmMDH对甲醇的Km为132 mmol/L[1,9],其对甲醇的亲和力比BsMDH更低.除此之外,还有来源于谷氨酸棒杆菌Corynebacterium glutamicum的MDH的Km也在97 mmol/L左右;来源于钩虫贪铜菌Cupriavidus necator N-1的MDH与BmMDH相似,对甲醇的亲和力在132 mmol/L左右,且对ACT不敏感;来源于解木糖赖氨酸芽孢杆菌Lysinibacillus xylanilyticus的MDH的Km在3 mmol/L左右,虽然对甲醇的亲和力相对较高,但其最适反应条件为pH9.5和55 ℃,不符合大肠杆菌的最适生长条件[10].研究表明,BsMDH易于在大肠杆菌中过量表达,已成功用于构建利用甲醇的工程菌株[11-12]或基于甲醇的胞外氧化还原体系[13]. ...
Cofactor engineering for advancing chemical biotechnology
1
2013
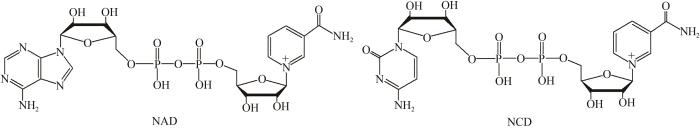
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Recent trends and novel concepts in cofactor-dependent biotransformations
1
2014
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
NAD(H) and NADP(H) redox couples and cellular energy metabolism
1
2018
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Rapid evolution of reversible denaturation and elevated melting temperature in a microbial haloalkane dehalogenase
1
2001
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Coping with complexity in metabolic engineering
1
2013
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Is simpler better? synthetic nicotinamide cofactor analogues for redox chemistry
1
2014
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Better than nature: nicotinamide biomimetics that outperform natural coenzymes
1
2016
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Applying non-canonical redox cofactors in fermentation processes
2
2020
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
... [21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Engineering a nicotinamide mononucleotide redox cofactor system for biocatalysis
1
2020
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Computational studies of the mechanism for proton and hydride transfer in liver alcohol dehydrogenase
1
2000
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Synthesis of NAD analogs to develop bioorthogonal redox system
1
2013
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Synthesis of 1,2,3-triazole moiety-containing NAD analogs and their potential as redox cofactors
0
2011
Synthesis of novel nicotinamide adenine dinucleotide (NAD) analogs and their coenzyme activities
1
2012
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Creation of bioorthogonal redox systems depending on nicotinamide flucytosine dinucleotide
3
2011
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
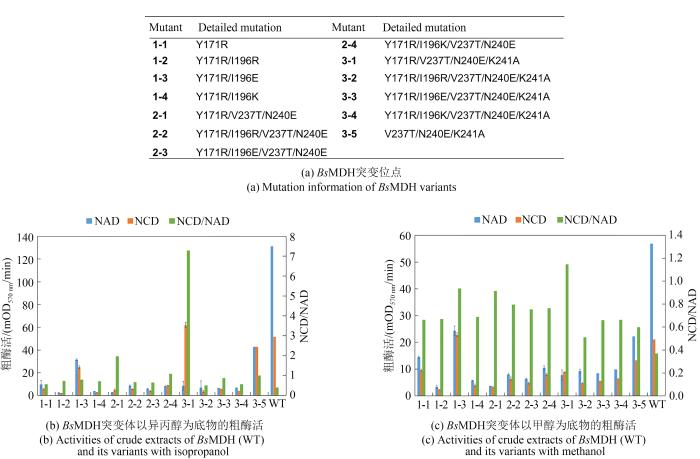
... 分析MDH的结构表明,V237和N240分别处于NAD腺嘌呤平面的两个方向.鉴于NAD和NCD的重要区别在于腺嘌呤比胞嘧啶基团空间更大,如果在腺嘌呤周围选择3个方向的位点进行突变,就可能更好地改变辅酶结合空腔大小,获得辅酶结合空腔缩小不利于NAD结合但适合NCD的突变体.通过分析MDH结构,并根据已有筛选经验[27],推测第3个方向的位点可能是Y171或I196. ...
... (1)改造其他氧化还原酶的辅酶偏好性.本文改造MDH辅酶偏好性的经验和流程,可能应用于其他NAD依赖型氧化还原酶如甲醛脱氢酶等.事实上,本文及前期改造苹果酸酶[27]、亚磷酸脱氢酶[45]和甲酸脱氢酶[46]的系列研究表明,在结构分析和半理性设计指导下,在辅酶结合空腔选择位点,构建突变体文库并筛选,并最终得到具有高NCD偏好性的突变体,通常均包含一些新引入的大侧链极性氨基酸突变.本研究的中后期,采用单位点突变方法构建13种突变体的实验,正是基于上述经验.因此,预计未来将其他NAD依赖型氧化还原酶改造为NCD偏好性的研究进程将变得更加有效和精准. ...
Synthetic cofactor-linked metabolic circuits for selective energy transfer
3
2017
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
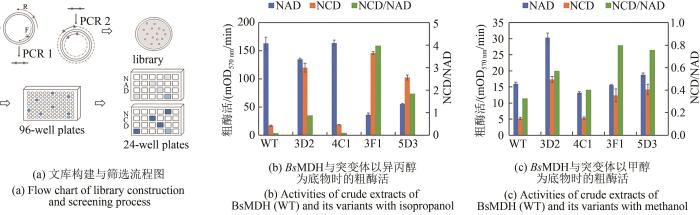
... 取上清粗酶液10 μL,在酶标板上分别进行与NAD和NCD的反应,在570 nm下测定光密度变化[28].测定结果以每分钟光密度的变化表示粗酶活(mOD570/min).每个样品3个平行.体系为HEPES(pH7.5)50 mmol/L,NAD、NCD的浓度根据酶对辅因子的活性而调整,异丙醇13 mmol/L或甲醇50 mmol/L,MTT 400 μmol/L,PES 1 mmol/L,粗酶液10 μL.鉴于MDH以异丙醇为底物时催化活性更高[32],在MDH活性筛选时,均首先以异丙醇为底物,再以甲醇进行复筛和验证. ...
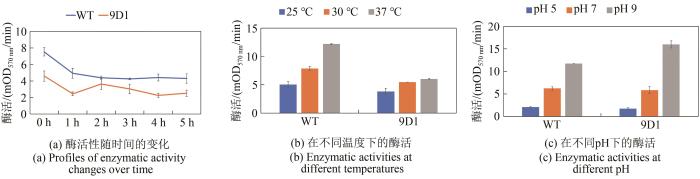
... 酶循环法测定MDH突变体的动力学,检测条件为25 ℃,检测波长570 nm[40].酶活测定体系:HEPES(pH7.5)50 mmol/L,甲醇800 mmol/L,MTT 400 μmol/L,PES 1 mmol/L,NAD或NCD的浓度呈梯度变化,加超纯水至100 μL,每个样品3个平行.分别将各酶稀释不同的倍数用于NAD或NCD检测[28].利用Origin8.5拟合米氏方程.根据kcat=Vm/[E]([E]为酶浓度)计算kcat. ...
Non-natural cofactor and formate-driven reductive carboxylation of pyruvate
2
2020
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
... (2)构建基于甲醇的新型氧化还原催化体系.甲醇可分3步逐级生物氧化为CO2,每步反应均有相应辅酶依赖型氧化还原酶从而产生计量的还原型辅酶.本研究得到NCD偏好型MDH将甲醇氧化为甲醛,而此前的研究已获得NCD偏好型甲酸脱氢酶[29,46].未来如果将甲醛脱氢酶的辅酶偏好性也予以改造并获得NCD偏好型突变体,即可构建以甲醇为还原性底物的NCDH供给体系,用于选择性为特定代谢反应或途径提供还原力.本课题组认为,这种基于甲醇的NCDH供给体系,与此前构建的基于亚磷酸[7]和甲酸[8]的体系相比,更加绿色并具有更好的经济性. ...
The significance of aspartate on NAD(H) biosynthesis and ABE fermentation in Clostridium acetobutylicum ATCC 824
1
2019
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
烟酰胺类辅酶依赖型氧化还原酶的辅酶偏好性改造及其在合成生物学中的应用
1
2020
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Protein engineering of nicotinamide coenzyme-dependent oxidoreductases for coenzyme preference and its application in synthetic biology
1
2020
... NAD及其还原型NADH是胞内重要的辅因子,广泛参与氧化还原代谢反应[14].NAD(H)还可以参与其他生物学过程如细胞繁殖、生长、分化和凋亡等[15].调控胞内NAD(H)水平是代谢工程及合成生物研究中改善宿主性能的常用手段,获得过大量成功案例[16].然而,NAD(H)水平扰动可对胞内代谢反应产生复杂且难以预测的影响[17-18].利用NAD类似物,可提高催化效率、促进辅因子循环再生或提高生物催化反应[19-20].虽然一些结构简化的NAD类似物可用于体外酶催化,但在胞内应用需特别考虑生物兼容性,如氧化还原电位、溶解性以及结构单元是否易于生物合成等[21].此外,也有研究尝试将NAD生物合成前体如烟酰胺单核苷酸(NMN)和化学修饰的NAD类似物用做非天然氧化还原辅酶[22-23].本课题组早期合成了一系列具有生物活性的NAD类似物[24-26],其中烟酰胺胞嘧啶二核苷酸(NCD,图1)与NAD在结构上存在一个碱基差异.以NAD依赖型苹果酸酶为模型,改造筛选获得了系列偏好NCD的苹果酸酶突变体[27],通过改造亚磷酸脱氢酶和甲酸脱氢酶的辅酶结合口袋,均得到了偏好NCD的突变体,并成功构建了NCD介导的、正交于内源氧化还原辅酶的代谢线路[28-29].最近,也有研究尝试构建基于NMN[30]及其他烟酰胺衍生物[21]的氧化还原催化体系,并取得了有价值的成果[31].这些研究不断丰富氧化还原催化元件,对合成生物学领域发展具有重要意义. ...
Characterization of the substrate scope of an alcohol dehydrogenase commonly used as methanol dehydrogenase
2
2019
... 构建文库及蛋白质表达及纯化时所用宿主为大肠杆菌BL21(DE3).密码子优化的BsMDH编码基因[8]由Synbio Technologies合成,并已克隆在表达质粒pTrc99K-MDH[32]. ...
... 取上清粗酶液10 μL,在酶标板上分别进行与NAD和NCD的反应,在570 nm下测定光密度变化[28].测定结果以每分钟光密度的变化表示粗酶活(mOD570/min).每个样品3个平行.体系为HEPES(pH7.5)50 mmol/L,NAD、NCD的浓度根据酶对辅因子的活性而调整,异丙醇13 mmol/L或甲醇50 mmol/L,MTT 400 μmol/L,PES 1 mmol/L,粗酶液10 μL.鉴于MDH以异丙醇为底物时催化活性更高[32],在MDH活性筛选时,均首先以异丙醇为底物,再以甲醇进行复筛和验证. ...
SWISS-MODEL: homology modelling of protein structures and complexes
1
2018
... 以序列相似度58%的醇脱氢酶(PDB ID:3s2e)的晶体结构为模板,用蛋白质结构同源性模型服务器(SWISS-MODEL)模拟BsMDH的结构[33],用软件PyMOL分析结构确定突变氨基酸残基的空间位置并作图[34].用MOE进行分子对接和结构分析.分子对接:BsMDH为受体,NCD为配体,NAD空腔为配体结合位点,对接选择默认方法,其中放置方法为Triangle Matcher,优化方法为Rigid Recepter,打分方法为London dG和GbBVI/WSA dG,pose值分别设置为300和50,虚拟筛选设计方法选择Residue Scan,筛选方法选择LowMode,突变位点选择1个. ...
Computer-aided drug design platform using PyMOL
1
2011
... 以序列相似度58%的醇脱氢酶(PDB ID:3s2e)的晶体结构为模板,用蛋白质结构同源性模型服务器(SWISS-MODEL)模拟BsMDH的结构[33],用软件PyMOL分析结构确定突变氨基酸残基的空间位置并作图[34].用MOE进行分子对接和结构分析.分子对接:BsMDH为受体,NCD为配体,NAD空腔为配体结合位点,对接选择默认方法,其中放置方法为Triangle Matcher,优化方法为Rigid Recepter,打分方法为London dG和GbBVI/WSA dG,pose值分别设置为300和50,虚拟筛选设计方法选择Residue Scan,筛选方法选择LowMode,突变位点选择1个. ...
Active site engineering of the epoxide hydrolase from Agrobacterium radiobacter AD1 to enhance aerobic mineralization of cis-1,2-dichloroethylene in cells expressing an evolved toluene ortho-monooxygenase
1
2004
... 利用96孔板的筛选技术构建饱和突变文库[35],由于密码子的简并性,选择NNK(N为A,G,T,C;K为G,T)代替原来的3个碱基覆盖所有可能的氨基酸,即用32个密码子可代表20种氨基酸.使用不依赖于限制性内切酶的克隆(RF克隆)方法[36]构建文库,对于单位点突变,设计具有简并密码子NNK的引物,然后进行RF克隆;对于多位点突变,首先扩增具有简并密码子的片段,然后进行RF克隆.构建文库时,以携带野生型MDH的质粒用作模板,将PCR产物在37 °C用DpnI处理2 h,然后将其转化进大肠杆菌BL21(DE3)细胞中,待转化子长出. ...
RF cloning: A restriction-free method for inserting target genes into plasmids
1
2006
... 利用96孔板的筛选技术构建饱和突变文库[35],由于密码子的简并性,选择NNK(N为A,G,T,C;K为G,T)代替原来的3个碱基覆盖所有可能的氨基酸,即用32个密码子可代表20种氨基酸.使用不依赖于限制性内切酶的克隆(RF克隆)方法[36]构建文库,对于单位点突变,设计具有简并密码子NNK的引物,然后进行RF克隆;对于多位点突变,首先扩增具有简并密码子的片段,然后进行RF克隆.构建文库时,以携带野生型MDH的质粒用作模板,将PCR产物在37 °C用DpnI处理2 h,然后将其转化进大肠杆菌BL21(DE3)细胞中,待转化子长出. ...
Site-saturated mutagenesis of histidine 234 of Saccharomyces cerevisiae oxidosqualene-lanosterol cyclase demonstrates dual functions in cyclization and rearrangement reactions
1
2006
... 为提高获得最优结果的概率,构建在每个位点都覆盖所有氨基酸的突变体文库,然后进行筛选[37-38].利用NBT-PMS偶联用方法,根据570 nm光吸收变化测定甲醇脱氢酶的活性[39-40].按文献方法估算拟筛选的转化子数目[41],保证覆盖相应文库理论值的95%. ...
Large-scale, saturating insertional mutagenesis of the mouse genome
1
2007
... 为提高获得最优结果的概率,构建在每个位点都覆盖所有氨基酸的突变体文库,然后进行筛选[37-38].利用NBT-PMS偶联用方法,根据570 nm光吸收变化测定甲醇脱氢酶的活性[39-40].按文献方法估算拟筛选的转化子数目[41],保证覆盖相应文库理论值的95%. ...
Guided evolution of enzymes with new substrate specificities
1
1996
... 为提高获得最优结果的概率,构建在每个位点都覆盖所有氨基酸的突变体文库,然后进行筛选[37-38].利用NBT-PMS偶联用方法,根据570 nm光吸收变化测定甲醇脱氢酶的活性[39-40].按文献方法估算拟筛选的转化子数目[41],保证覆盖相应文库理论值的95%. ...
Identification of UshA as a major enzyme for NAD degradation in Escherichia coli
2
2014
... 为提高获得最优结果的概率,构建在每个位点都覆盖所有氨基酸的突变体文库,然后进行筛选[37-38].利用NBT-PMS偶联用方法,根据570 nm光吸收变化测定甲醇脱氢酶的活性[39-40].按文献方法估算拟筛选的转化子数目[41],保证覆盖相应文库理论值的95%. ...
... 酶循环法测定MDH突变体的动力学,检测条件为25 ℃,检测波长570 nm[40].酶活测定体系:HEPES(pH7.5)50 mmol/L,甲醇800 mmol/L,MTT 400 μmol/L,PES 1 mmol/L,NAD或NCD的浓度呈梯度变化,加超纯水至100 μL,每个样品3个平行.分别将各酶稀释不同的倍数用于NAD或NCD检测[28].利用Origin8.5拟合米氏方程.根据kcat=Vm/[E]([E]为酶浓度)计算kcat. ...
Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes
1
2007
... 为提高获得最优结果的概率,构建在每个位点都覆盖所有氨基酸的突变体文库,然后进行筛选[37-38].利用NBT-PMS偶联用方法,根据570 nm光吸收变化测定甲醇脱氢酶的活性[39-40].按文献方法估算拟筛选的转化子数目[41],保证覆盖相应文库理论值的95%. ...
High resolution structures of holo and apo formate dehydrogense
1
1994
... 对MDH位点的选择主要依赖于分析晶体结构中氨基酸残基与NAD腺嘌呤的空间相对位置[42],再结合晶体中氨基酸残基与NAD腺嘌呤相互作用分析,采用迭代饱和突变和组合突变等方法对突变文库进行半理性设计[43-44]. ...
Directed evolution of enantioselective enzymes: Iterative cycles of CASTing for probing protein-sequence space
1
2006
... 对MDH位点的选择主要依赖于分析晶体结构中氨基酸残基与NAD腺嘌呤的空间相对位置[42],再结合晶体中氨基酸残基与NAD腺嘌呤相互作用分析,采用迭代饱和突变和组合突变等方法对突变文库进行半理性设计[43-44]. ...
Catalytic asymmetric reduction of difficult-to-reduce ketones: triple-code saturation mutagenesis of an alcohol dehydrogenase
1
2016
... 对MDH位点的选择主要依赖于分析晶体结构中氨基酸残基与NAD腺嘌呤的空间相对位置[42],再结合晶体中氨基酸残基与NAD腺嘌呤相互作用分析,采用迭代饱和突变和组合突变等方法对突变文库进行半理性设计[43-44]. ...
Structural insights into phosphite dehydrogenase variants favoring a non-natural redox cofactor
2
2019
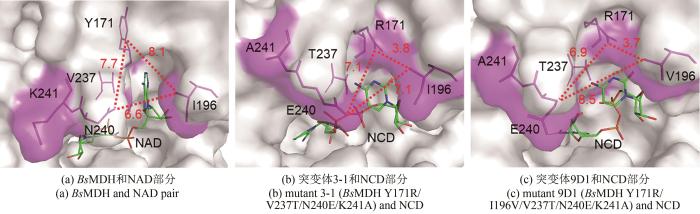
... 对BsMDH和突变体3-1、9D1进行分子对接,分别对接NAD和NCD,并分析辅酶结合空腔大小.从图6可以看出,BsMDH中Y171、I196和N240形成的空腔远大于突变体3-1和9D1中相应残基形成的空腔.BsMDH中Y171和I196之间的距离为0.81 nm,而突变体3-1中R171和I196之间的距离缩短到0.38 nm,突变体9D1中R171和V196之间的距离为0.37 nm.此外,Y171R突变是辅酶结合空腔变小的主要原因.由于NAD的腺嘌呤结构在空间上大于NCD的胞嘧啶结构,突变体辅酶结合空腔变小导致难以结合NAD,而有利于结合NCD.初步说明辅酶结合空腔变小是MDH突变体偏好非天然辅酶NCD的主要原因.这较好地吻合了亚磷酸脱氢酶辅酶偏好性改造研究中通过突变体-NCD复合物晶体结构分析观察到的现象[45]. ...
... (1)改造其他氧化还原酶的辅酶偏好性.本文改造MDH辅酶偏好性的经验和流程,可能应用于其他NAD依赖型氧化还原酶如甲醛脱氢酶等.事实上,本文及前期改造苹果酸酶[27]、亚磷酸脱氢酶[45]和甲酸脱氢酶[46]的系列研究表明,在结构分析和半理性设计指导下,在辅酶结合空腔选择位点,构建突变体文库并筛选,并最终得到具有高NCD偏好性的突变体,通常均包含一些新引入的大侧链极性氨基酸突变.本研究的中后期,采用单位点突变方法构建13种突变体的实验,正是基于上述经验.因此,预计未来将其他NAD依赖型氧化还原酶改造为NCD偏好性的研究进程将变得更加有效和精准. ...
Structure-guided design of formate dehydrogenase for regeneration of a non-natural redox cofactor
2
2020
... (1)改造其他氧化还原酶的辅酶偏好性.本文改造MDH辅酶偏好性的经验和流程,可能应用于其他NAD依赖型氧化还原酶如甲醛脱氢酶等.事实上,本文及前期改造苹果酸酶[27]、亚磷酸脱氢酶[45]和甲酸脱氢酶[46]的系列研究表明,在结构分析和半理性设计指导下,在辅酶结合空腔选择位点,构建突变体文库并筛选,并最终得到具有高NCD偏好性的突变体,通常均包含一些新引入的大侧链极性氨基酸突变.本研究的中后期,采用单位点突变方法构建13种突变体的实验,正是基于上述经验.因此,预计未来将其他NAD依赖型氧化还原酶改造为NCD偏好性的研究进程将变得更加有效和精准. ...
... (2)构建基于甲醇的新型氧化还原催化体系.甲醇可分3步逐级生物氧化为CO2,每步反应均有相应辅酶依赖型氧化还原酶从而产生计量的还原型辅酶.本研究得到NCD偏好型MDH将甲醇氧化为甲醛,而此前的研究已获得NCD偏好型甲酸脱氢酶[29,46].未来如果将甲醛脱氢酶的辅酶偏好性也予以改造并获得NCD偏好型突变体,即可构建以甲醇为还原性底物的NCDH供给体系,用于选择性为特定代谢反应或途径提供还原力.本课题组认为,这种基于甲醇的NCDH供给体系,与此前构建的基于亚磷酸[7]和甲酸[8]的体系相比,更加绿色并具有更好的经济性. ...