Harnessing diverse transcriptional regulators for natural product discovery in fungi
2
2020
... 通过测序和注释,发现真菌基因组中存在大量参与非核糖体多肽天然产物生物合成的基因簇[1],具有合成结构多样化合物的巨大潜力[2].大多数真菌非核糖体多肽合成酶(nonribosomal polypeptide synthetase,NRPS)包括多个由腺苷酰化(adenylation,A)、巯基化(thiolation,T)和缩合(condensation,C)等结构域组成的模块.在生物合成的过程中,腺苷酰化结构域先将氨基酸或羟基酸前体单元加载到巯基化结构域上,然后缩合结构域缩合前体单元和中间产物合成非核糖体多肽[3].除了这3个核心结构域,非核糖体多肽合成酶还可能包含修饰前体单元或中间产物甲基化、差向异构化的结构域,以及终止延伸和释放产物、末端缩合等功能的结构域,从而使合成的产物结构更加复杂多样. ...
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
Atlas of nonribosomal peptide and polyketide biosynthetic pathways reveals common occurrence of nonmodular enzymes
1
2014
... 通过测序和注释,发现真菌基因组中存在大量参与非核糖体多肽天然产物生物合成的基因簇[1],具有合成结构多样化合物的巨大潜力[2].大多数真菌非核糖体多肽合成酶(nonribosomal polypeptide synthetase,NRPS)包括多个由腺苷酰化(adenylation,A)、巯基化(thiolation,T)和缩合(condensation,C)等结构域组成的模块.在生物合成的过程中,腺苷酰化结构域先将氨基酸或羟基酸前体单元加载到巯基化结构域上,然后缩合结构域缩合前体单元和中间产物合成非核糖体多肽[3].除了这3个核心结构域,非核糖体多肽合成酶还可能包含修饰前体单元或中间产物甲基化、差向异构化的结构域,以及终止延伸和释放产物、末端缩合等功能的结构域,从而使合成的产物结构更加复杂多样. ...
Secondary metabolites from entomopathogenic Hypocrealean fungi
3
2010
... 通过测序和注释,发现真菌基因组中存在大量参与非核糖体多肽天然产物生物合成的基因簇[1],具有合成结构多样化合物的巨大潜力[2].大多数真菌非核糖体多肽合成酶(nonribosomal polypeptide synthetase,NRPS)包括多个由腺苷酰化(adenylation,A)、巯基化(thiolation,T)和缩合(condensation,C)等结构域组成的模块.在生物合成的过程中,腺苷酰化结构域先将氨基酸或羟基酸前体单元加载到巯基化结构域上,然后缩合结构域缩合前体单元和中间产物合成非核糖体多肽[3].除了这3个核心结构域,非核糖体多肽合成酶还可能包含修饰前体单元或中间产物甲基化、差向异构化的结构域,以及终止延伸和释放产物、末端缩合等功能的结构域,从而使合成的产物结构更加复杂多样. ...
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
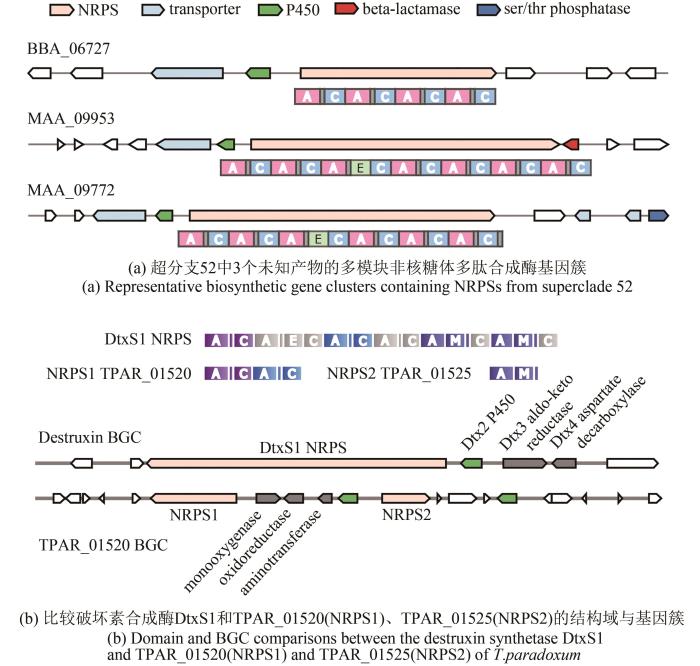
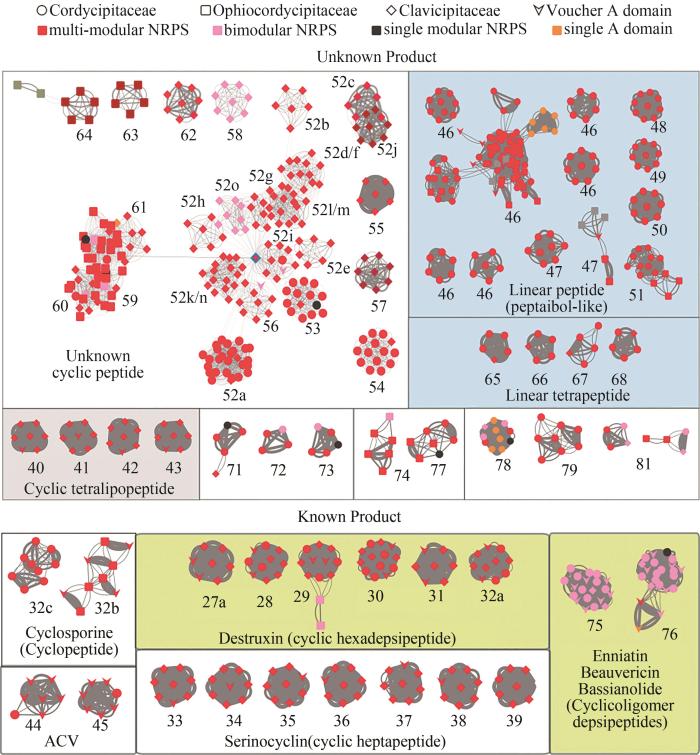
... 这些多模块非核糖体多肽合成酶中,绝大多数的产物是未知的.如图1所示,第52~64分支的非核糖体多肽合成酶形成了一个较大的超级分支(super clade).根据氨基酸序列相似性,这些合成酶的最后一个结构域属于环肽类的终端缩合结构域,可以通过分子内环化释放延伸的肽链,因此推测其产物是环肽类化合物.其中,52~60分支可以进一步划分为3种含有4个以上模块的非核糖体多肽合成酶,其代表为B. bassiana的BBA_06727、M. robertsii的MAA_09953和MAA_09772(图1).通过系统发育分析,这些非核糖体多肽合成酶曾被归为“昆虫病原菌特有”的进化分支[3-4].与BBA_06727相似的蛋白有13个,结构域组成均为(AT)-(CAT)-(CAT)-(CAT)-(CT),它们的第1个腺苷酰化结构域归于第53个分支,第3个腺苷酰化结构域归于第54分支,第2个和第4个腺苷酰化结构域均归于分支52a.同一个非核糖体多肽合成酶中的两个腺苷酰化结构域聚类在一起通常表明它们来源于进化上的复制事件.这一类非核糖体多肽合成酶所在的基因簇中还含有编码细胞色素P450单加氧酶和转运蛋白的基因[图2(a)].BBA_06727的转录在昆虫感染期间被激活[27],表明可能在昆虫致病过程中起作用.52b~52h亚分支和第55分支的非核糖体多肽合成酶(如MAA_09953)存在于6种绿僵菌属真菌基因组中,其结构域组成均为(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CAT)-(CT),所在基因簇中还包含编码细胞色素P450单加氧酶、β-内酰胺酶和转运蛋白的基因.以MAA_09772为代表的非核糖体多肽合成酶在8种绿僵菌属真菌中十分保守,具有(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CTT)的结构域组成,其腺苷酰化结构域归于分支59、亚分支52i~52n、分支60,相应的合成基因簇包含细胞色素P450单加氧酶、Ser/Thr蛋白磷酸酶和两个转运蛋白的基因.亚分支52o和分支58的腺苷酰化结构域也来源于相同的非核糖体多肽合成酶,其结构域的组成为(AT)-(CATE)-(CT)-(CT),基因簇含有编码细胞色素P450单加氧酶、两种氨基转移酶、脱羧酶、双加氧酶和含有腺苷酰化结构域的连接酶/合成酶的基因.值得注意的是,超级分支52中几乎所有的分支都是通过52i中的一个节点连接的,这个节点对应于M.rileyi基因组中NOR_07696的第2个腺苷酰化结构域(介数betweenness为0.826),表明这个腺苷酰化结构域与共同的祖先最为相似.分支59~61中来自于线虫草科(Ophiocordycipitaceae)和麦角菌科(Clavicipitaceae)的非核糖体多肽合成酶也通过NOR_07696相连. ...
Discovering the secondary metabolite potential encoded within entomopathogenic fungi
3
2014
... 腺苷酰化和缩合结构域均已成功用于非核糖体多肽合成酶系统发育研究[4, 21-26].本研究在网络构建和聚类分析腺苷酰化结构域的基础上,分析了部分非核糖体多肽合成酶的模块和结构域组成,以及所在的基因簇特征.研究结果显示肉座菌目虫生真菌的基因组中广泛存在功能未知的非核糖体多肽合成酶家族,即使在亲缘关系十分接近的种属之间,它们在基因或基因簇水平上也存在高度变异性,表明肉座菌目虫生真菌是合成新型非核糖体多肽类天然产物的潜在资源. ...
... 肉座菌目虫生真菌基因组预测的2440个腺苷酰化结构域中,有1056个来自于多模块的非核糖体多肽合成酶(图1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4, 24],是发现新型活性产物的宝贵资源. ...
... 这些多模块非核糖体多肽合成酶中,绝大多数的产物是未知的.如图1所示,第52~64分支的非核糖体多肽合成酶形成了一个较大的超级分支(super clade).根据氨基酸序列相似性,这些合成酶的最后一个结构域属于环肽类的终端缩合结构域,可以通过分子内环化释放延伸的肽链,因此推测其产物是环肽类化合物.其中,52~60分支可以进一步划分为3种含有4个以上模块的非核糖体多肽合成酶,其代表为B. bassiana的BBA_06727、M. robertsii的MAA_09953和MAA_09772(图1).通过系统发育分析,这些非核糖体多肽合成酶曾被归为“昆虫病原菌特有”的进化分支[3-4].与BBA_06727相似的蛋白有13个,结构域组成均为(AT)-(CAT)-(CAT)-(CAT)-(CT),它们的第1个腺苷酰化结构域归于第53个分支,第3个腺苷酰化结构域归于第54分支,第2个和第4个腺苷酰化结构域均归于分支52a.同一个非核糖体多肽合成酶中的两个腺苷酰化结构域聚类在一起通常表明它们来源于进化上的复制事件.这一类非核糖体多肽合成酶所在的基因簇中还含有编码细胞色素P450单加氧酶和转运蛋白的基因[图2(a)].BBA_06727的转录在昆虫感染期间被激活[27],表明可能在昆虫致病过程中起作用.52b~52h亚分支和第55分支的非核糖体多肽合成酶(如MAA_09953)存在于6种绿僵菌属真菌基因组中,其结构域组成均为(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CAT)-(CT),所在基因簇中还包含编码细胞色素P450单加氧酶、β-内酰胺酶和转运蛋白的基因.以MAA_09772为代表的非核糖体多肽合成酶在8种绿僵菌属真菌中十分保守,具有(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CTT)的结构域组成,其腺苷酰化结构域归于分支59、亚分支52i~52n、分支60,相应的合成基因簇包含细胞色素P450单加氧酶、Ser/Thr蛋白磷酸酶和两个转运蛋白的基因.亚分支52o和分支58的腺苷酰化结构域也来源于相同的非核糖体多肽合成酶,其结构域的组成为(AT)-(CATE)-(CT)-(CT),基因簇含有编码细胞色素P450单加氧酶、两种氨基转移酶、脱羧酶、双加氧酶和含有腺苷酰化结构域的连接酶/合成酶的基因.值得注意的是,超级分支52中几乎所有的分支都是通过52i中的一个节点连接的,这个节点对应于M.rileyi基因组中NOR_07696的第2个腺苷酰化结构域(介数betweenness为0.826),表明这个腺苷酰化结构域与共同的祖先最为相似.分支59~61中来自于线虫草科(Ophiocordycipitaceae)和麦角菌科(Clavicipitaceae)的非核糖体多肽合成酶也通过NOR_07696相连. ...
Symbionts, a promising source of bioactive natural products
0
2015
Natural products from microbes associated with insects
0
2016
Molecular interactions between entomopathogenic fungi (Hypocreales) and their insect host: perspectives from stressful cuticle and hemolymph battlefields and the potential of dual RNA sequencing for future studies
0
2018
The genus Cordyceps: an extensive review of its traditional uses, phytochemistry and pharmacology
1
2018
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
Unveiling the biosynthetic puzzle of destruxins in Metarhizium species
4
2012
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
... 肉座菌目虫生真菌基因组预测的2440个腺苷酰化结构域中,有1056个来自于多模块的非核糖体多肽合成酶(图1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4, 24],是发现新型活性产物的宝贵资源. ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. The nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. Boxed groups show A domains from the group of enzymes that also contain DtxS1, or superclades that are formed by closely related A domain clades such as clades #52~64 (see text for details). Sub-clades of A domains are indicated with a number-letter combination, for example 52a. NRPSs of the BGCs that contain the voucher A domains include: #27a~32a, destruxin synthetase DtxS1[9]; #32b, cyclosporine synthetase[29]; #33~39, serinocyclin synthetase[13-14]; #44~45, ACVS: N-(5-amino-5-carboxypentanoyl)-L-cysteinyl-D-valine synthetase in penicillin/cephalosporin biosynthesis; #46~51, Tex1: a peptaibol synthetase from Trichoderma virens[30]; #75~76, beauvericin and bassianolide synthetase[11-12].) ...
... 除此之外,一些分支包含产物已知的非核糖体多肽合成酶,具有相同的结构域组成,所在的基因簇也高度相似,可能产生与已知化合物结构类似的产物.其中,分支75~76包含白僵菌素/白僵菌环四肽合成酶的腺苷酰化结构域[11, 31],分支27~32和33~39则分别包含破坏素[9-10]和serinocyclin[13]合成酶的腺苷酰化结构域(图1),说明这些分支中的菌株具有合成杀虫化合物的潜力. ...
Genetic basis of destruxin production in the entomopathogen Metarhizium robertsii
1
2012
... 除此之外,一些分支包含产物已知的非核糖体多肽合成酶,具有相同的结构域组成,所在的基因簇也高度相似,可能产生与已知化合物结构类似的产物.其中,分支75~76包含白僵菌素/白僵菌环四肽合成酶的腺苷酰化结构域[11, 31],分支27~32和33~39则分别包含破坏素[9-10]和serinocyclin[13]合成酶的腺苷酰化结构域(图1),说明这些分支中的菌株具有合成杀虫化合物的潜力. ...
Biosynthesis of the cyclooligomer depsipeptide beauvericin, a virulence factor of the entomopathogenic fungus Beauveria bassiana
3
2008
... 肉座菌目虫生真菌基因组预测的2440个腺苷酰化结构域中,有1056个来自于多模块的非核糖体多肽合成酶(图1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4, 24],是发现新型活性产物的宝贵资源. ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. The nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. Boxed groups show A domains from the group of enzymes that also contain DtxS1, or superclades that are formed by closely related A domain clades such as clades #52~64 (see text for details). Sub-clades of A domains are indicated with a number-letter combination, for example 52a. NRPSs of the BGCs that contain the voucher A domains include: #27a~32a, destruxin synthetase DtxS1[9]; #32b, cyclosporine synthetase[29]; #33~39, serinocyclin synthetase[13-14]; #44~45, ACVS: N-(5-amino-5-carboxypentanoyl)-L-cysteinyl-D-valine synthetase in penicillin/cephalosporin biosynthesis; #46~51, Tex1: a peptaibol synthetase from Trichoderma virens[30]; #75~76, beauvericin and bassianolide synthetase[11-12].) ...
... 除此之外,一些分支包含产物已知的非核糖体多肽合成酶,具有相同的结构域组成,所在的基因簇也高度相似,可能产生与已知化合物结构类似的产物.其中,分支75~76包含白僵菌素/白僵菌环四肽合成酶的腺苷酰化结构域[11, 31],分支27~32和33~39则分别包含破坏素[9-10]和serinocyclin[13]合成酶的腺苷酰化结构域(图1),说明这些分支中的菌株具有合成杀虫化合物的潜力. ...
Biosynthesis of the cyclooligomer depsipeptide bassianolide, an insecticidal virulence factor of Beauveria bassiana
1
2009
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. The nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. Boxed groups show A domains from the group of enzymes that also contain DtxS1, or superclades that are formed by closely related A domain clades such as clades #52~64 (see text for details). Sub-clades of A domains are indicated with a number-letter combination, for example 52a. NRPSs of the BGCs that contain the voucher A domains include: #27a~32a, destruxin synthetase DtxS1[9]; #32b, cyclosporine synthetase[29]; #33~39, serinocyclin synthetase[13-14]; #44~45, ACVS: N-(5-amino-5-carboxypentanoyl)-L-cysteinyl-D-valine synthetase in penicillin/cephalosporin biosynthesis; #46~51, Tex1: a peptaibol synthetase from Trichoderma virens[30]; #75~76, beauvericin and bassianolide synthetase[11-12].) ...
Agrobacterium-mediated disruption of a nonribosomal peptide synthetase gene in the invertebrate pathogen Metarhizium anisopliae reveals a peptide spore factor
3
2008
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. The nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. Boxed groups show A domains from the group of enzymes that also contain DtxS1, or superclades that are formed by closely related A domain clades such as clades #52~64 (see text for details). Sub-clades of A domains are indicated with a number-letter combination, for example 52a. NRPSs of the BGCs that contain the voucher A domains include: #27a~32a, destruxin synthetase DtxS1[9]; #32b, cyclosporine synthetase[29]; #33~39, serinocyclin synthetase[13-14]; #44~45, ACVS: N-(5-amino-5-carboxypentanoyl)-L-cysteinyl-D-valine synthetase in penicillin/cephalosporin biosynthesis; #46~51, Tex1: a peptaibol synthetase from Trichoderma virens[30]; #75~76, beauvericin and bassianolide synthetase[11-12].) ...
... 除此之外,一些分支包含产物已知的非核糖体多肽合成酶,具有相同的结构域组成,所在的基因簇也高度相似,可能产生与已知化合物结构类似的产物.其中,分支75~76包含白僵菌素/白僵菌环四肽合成酶的腺苷酰化结构域[11, 31],分支27~32和33~39则分别包含破坏素[9-10]和serinocyclin[13]合成酶的腺苷酰化结构域(图1),说明这些分支中的菌株具有合成杀虫化合物的潜力. ...
Secondary metabolite gene clusters in the entomopathogen fungus Metarhizium anisopliae: genome identification and patterns of expression in a cuticle infection model
3
2016
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
... 肉座菌目虫生真菌基因组预测的2440个腺苷酰化结构域中,有1056个来自于多模块的非核糖体多肽合成酶(图1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4, 24],是发现新型活性产物的宝贵资源. ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. The nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. Boxed groups show A domains from the group of enzymes that also contain DtxS1, or superclades that are formed by closely related A domain clades such as clades #52~64 (see text for details). Sub-clades of A domains are indicated with a number-letter combination, for example 52a. NRPSs of the BGCs that contain the voucher A domains include: #27a~32a, destruxin synthetase DtxS1[9]; #32b, cyclosporine synthetase[29]; #33~39, serinocyclin synthetase[13-14]; #44~45, ACVS: N-(5-amino-5-carboxypentanoyl)-L-cysteinyl-D-valine synthetase in penicillin/cephalosporin biosynthesis; #46~51, Tex1: a peptaibol synthetase from Trichoderma virens[30]; #75~76, beauvericin and bassianolide synthetase[11-12].) ...
Gene expression during zombie ant biting behavior reflects the complexity underlying fungal parasitic behavioral manipulation
1
2015
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
Deletion of a global regulator LaeB leads to the discovery of novel polyketides in Aspergillus nidulans
1
2018
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
Discovery of microbial natural products by activation of silent biosynthetic gene clusters
1
2015
... 肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8].通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15].在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1, 16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力.如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题.因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇.准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件. ...
Exploring the sequence, function, and evolutionary space of protein superfamilies using sequence similarity networks and phylogenetic reconstructions
1
2019
... 序列相似性网络(sequence similarity network,SSN)能够通过同源蛋白之间成对的序列相似关系,自动化评估蛋白的分类,并从家族/亚家族之间的关系来研究特定蛋白的功能.在SSN中,每个蛋白用一个符号(“节点”)表示;如果两个节点的序列相似性超过指定阈值,则通过一条线(“边”)连接.已有多篇文章综述了SSN在蛋白质序列与功能研究中的成功应用[18-20]. ...
Revealing unexplored sequence-function space using sequence similarity networks
0
2018
The structure-function linkage database
1
2014
... 序列相似性网络(sequence similarity network,SSN)能够通过同源蛋白之间成对的序列相似关系,自动化评估蛋白的分类,并从家族/亚家族之间的关系来研究特定蛋白的功能.在SSN中,每个蛋白用一个符号(“节点”)表示;如果两个节点的序列相似性超过指定阈值,则通过一条线(“边”)连接.已有多篇文章综述了SSN在蛋白质序列与功能研究中的成功应用[18-20]. ...
Global analysis of biosynthetic gene clusters reveals vast potential of secondary metabolite production in Penicillium species
1
2017
... 腺苷酰化和缩合结构域均已成功用于非核糖体多肽合成酶系统发育研究[4, 21-26].本研究在网络构建和聚类分析腺苷酰化结构域的基础上,分析了部分非核糖体多肽合成酶的模块和结构域组成,以及所在的基因簇特征.研究结果显示肉座菌目虫生真菌的基因组中广泛存在功能未知的非核糖体多肽合成酶家族,即使在亲缘关系十分接近的种属之间,它们在基因或基因簇水平上也存在高度变异性,表明肉座菌目虫生真菌是合成新型非核糖体多肽类天然产物的潜在资源. ...
Diversity and evolution of secondary metabolism in the marine actinomycete genus Salinispora
0
2014
Phylogenetic study of polyketide synthases and nonribosomal peptide synthetases involved in the biosynthesis of mycotoxins
0
2013
Phylogenomics reveals subfamilies of fungal nonribosomal peptide synthetases and their evolutionary relationships
6
2010
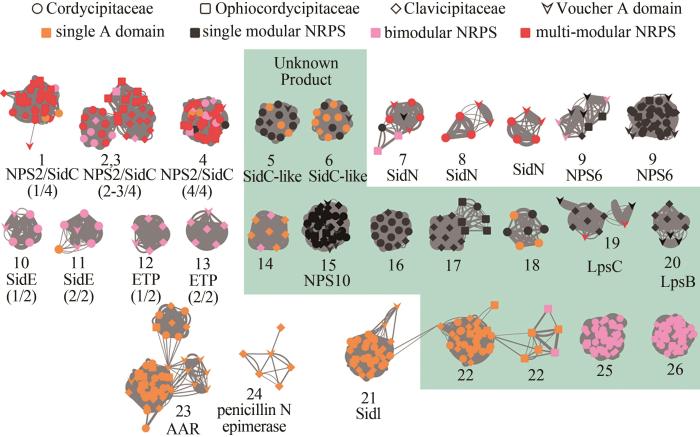
... 大多数非核糖体多肽合成酶具有多个腺苷酰化结构域,因此,利用对腺苷酰化结构域进行聚类分析来阐明合成酶的功能并预测产物结构变得十分复杂.在真菌聚酮合酶中,酮脂酰合酶(ketoacyl synthase,KS)结构域往往可以代表整个酶的特征;与此相对的,非核糖体多肽合成酶被视为具有不同系统发育特征的模块复合体,而腺苷酰化结构域的聚类则代表其所在模块的特征.收集到的2676个腺苷酰化结构域(包括302个已知功能的标签结构域)的氨基酸序列平均一致性为36.30%,其中2358个被成功地分为174个分支(不含聚酮合酶-非核糖体多肽合成酶杂合酶中的腺苷酰化结构域).由于数目较多,本文图中仅列出具有5个以上节点的分支簇,且排除了参与短链脂肪酸合成的酰基辅酶A连接酶(http://www.researchgate.net/publication/354887574_Table S1).由此产生的网络可将腺苷酰化结构域分为多模块、双模块、单模块和杂合的非核糖体多肽合成酶.这与先前报道的系统发育分析基本一致,即真菌非核糖体多肽合成酶分为两大分支:远古起源的单/双模块和新近起源的多/双模块[24, 27-28]. ...
... 肉座菌目虫生真菌基因组预测的2440个腺苷酰化结构域中,有1056个来自于多模块的非核糖体多肽合成酶(图1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4, 24],是发现新型活性产物的宝贵资源. ...
... 如图3所示,单/双模块的非核糖体多肽合成酶包括赖氨酸生物合成途径中还原α-氨基己二酸的L-氨基己二酸半醛脱氢酶(分支23)[24],参与形态发育但产物未知的ChNPS10合成酶(分支15)[40],产生聚硫二酮哌嗪的ChNPS11/ETP毒素合成酶(分支12和13)等[41],其合成的代谢物参与了真菌重要的生物过程,如性发育、生殖、分生孢子发育等,因此在基因组中也较为保守[24,32-39].其中,部分基因的生理功能已有研究,但其合成产物仍然未知. ...
... [24,32-39].其中,部分基因的生理功能已有研究,但其合成产物仍然未知. ...
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
Phylogenomic analysis of type I polyketide synthase genes in pathogenic and saprobic ascomycetes
0
2003
Phylogenetic analysis of condensation domains in NRPS sheds light on their functional evolution
1
2007
... 腺苷酰化和缩合结构域均已成功用于非核糖体多肽合成酶系统发育研究[4, 21-26].本研究在网络构建和聚类分析腺苷酰化结构域的基础上,分析了部分非核糖体多肽合成酶的模块和结构域组成,以及所在的基因簇特征.研究结果显示肉座菌目虫生真菌的基因组中广泛存在功能未知的非核糖体多肽合成酶家族,即使在亲缘关系十分接近的种属之间,它们在基因或基因簇水平上也存在高度变异性,表明肉座菌目虫生真菌是合成新型非核糖体多肽类天然产物的潜在资源. ...
The stress-responsive and host-oriented role of nonribosomal peptide synthetases in an entomopathogenic fungus, Beauveria bassiana
3
2017
... 大多数非核糖体多肽合成酶具有多个腺苷酰化结构域,因此,利用对腺苷酰化结构域进行聚类分析来阐明合成酶的功能并预测产物结构变得十分复杂.在真菌聚酮合酶中,酮脂酰合酶(ketoacyl synthase,KS)结构域往往可以代表整个酶的特征;与此相对的,非核糖体多肽合成酶被视为具有不同系统发育特征的模块复合体,而腺苷酰化结构域的聚类则代表其所在模块的特征.收集到的2676个腺苷酰化结构域(包括302个已知功能的标签结构域)的氨基酸序列平均一致性为36.30%,其中2358个被成功地分为174个分支(不含聚酮合酶-非核糖体多肽合成酶杂合酶中的腺苷酰化结构域).由于数目较多,本文图中仅列出具有5个以上节点的分支簇,且排除了参与短链脂肪酸合成的酰基辅酶A连接酶(http://www.researchgate.net/publication/354887574_Table S1).由此产生的网络可将腺苷酰化结构域分为多模块、双模块、单模块和杂合的非核糖体多肽合成酶.这与先前报道的系统发育分析基本一致,即真菌非核糖体多肽合成酶分为两大分支:远古起源的单/双模块和新近起源的多/双模块[24, 27-28]. ...
... 这些多模块非核糖体多肽合成酶中,绝大多数的产物是未知的.如图1所示,第52~64分支的非核糖体多肽合成酶形成了一个较大的超级分支(super clade).根据氨基酸序列相似性,这些合成酶的最后一个结构域属于环肽类的终端缩合结构域,可以通过分子内环化释放延伸的肽链,因此推测其产物是环肽类化合物.其中,52~60分支可以进一步划分为3种含有4个以上模块的非核糖体多肽合成酶,其代表为B. bassiana的BBA_06727、M. robertsii的MAA_09953和MAA_09772(图1).通过系统发育分析,这些非核糖体多肽合成酶曾被归为“昆虫病原菌特有”的进化分支[3-4].与BBA_06727相似的蛋白有13个,结构域组成均为(AT)-(CAT)-(CAT)-(CAT)-(CT),它们的第1个腺苷酰化结构域归于第53个分支,第3个腺苷酰化结构域归于第54分支,第2个和第4个腺苷酰化结构域均归于分支52a.同一个非核糖体多肽合成酶中的两个腺苷酰化结构域聚类在一起通常表明它们来源于进化上的复制事件.这一类非核糖体多肽合成酶所在的基因簇中还含有编码细胞色素P450单加氧酶和转运蛋白的基因[图2(a)].BBA_06727的转录在昆虫感染期间被激活[27],表明可能在昆虫致病过程中起作用.52b~52h亚分支和第55分支的非核糖体多肽合成酶(如MAA_09953)存在于6种绿僵菌属真菌基因组中,其结构域组成均为(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CAT)-(CT),所在基因簇中还包含编码细胞色素P450单加氧酶、β-内酰胺酶和转运蛋白的基因.以MAA_09772为代表的非核糖体多肽合成酶在8种绿僵菌属真菌中十分保守,具有(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CTT)的结构域组成,其腺苷酰化结构域归于分支59、亚分支52i~52n、分支60,相应的合成基因簇包含细胞色素P450单加氧酶、Ser/Thr蛋白磷酸酶和两个转运蛋白的基因.亚分支52o和分支58的腺苷酰化结构域也来源于相同的非核糖体多肽合成酶,其结构域的组成为(AT)-(CATE)-(CT)-(CT),基因簇含有编码细胞色素P450单加氧酶、两种氨基转移酶、脱羧酶、双加氧酶和含有腺苷酰化结构域的连接酶/合成酶的基因.值得注意的是,超级分支52中几乎所有的分支都是通过52i中的一个节点连接的,这个节点对应于M.rileyi基因组中NOR_07696的第2个腺苷酰化结构域(介数betweenness为0.826),表明这个腺苷酰化结构域与共同的祖先最为相似.分支59~61中来自于线虫草科(Ophiocordycipitaceae)和麦角菌科(Clavicipitaceae)的非核糖体多肽合成酶也通过NOR_07696相连. ...
... 铁载体是一种低分子量的铁螯合物,可分为胞外和胞内两种类型,因与病原真菌的致病性相关而受到广泛关注[37].铁载体合成酶具有保守的结构域,合成产物的结构和生物学功能相似,在各种营养模式的真菌中广泛分布,包括动植物病原菌和腐生菌.铁载体合成酶是多模块的非核糖体多肽合成酶,在系统发育中属于较新的分支,然而由于其序列和功能的保守性,将它们与单/双模块的非核糖体多肽合成酶一起分析.大部分肉座菌目虫生真菌的基因组中都有铁载体合成酶NPS2/SidC(分支1~4)[36]、NPS6(分支9)[39]、SidN(分支7和8)[38]和/或SidI(分支21)[37]的编码基因.值得注意的是,分支5和6的腺苷酰化结构域与NPS2/SidC的第1个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在.这表明该分支的类SidC非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能.分支10~11的双模块非核糖体多肽合成酶与人类病原菌A. fumigatus的SidE相似[42].SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
Genome sequence and comparative analysis of clavicipitaceous insect-pathogenic fungus Aschersonia badia with Metarhizium spp
1
2016
... 大多数非核糖体多肽合成酶具有多个腺苷酰化结构域,因此,利用对腺苷酰化结构域进行聚类分析来阐明合成酶的功能并预测产物结构变得十分复杂.在真菌聚酮合酶中,酮脂酰合酶(ketoacyl synthase,KS)结构域往往可以代表整个酶的特征;与此相对的,非核糖体多肽合成酶被视为具有不同系统发育特征的模块复合体,而腺苷酰化结构域的聚类则代表其所在模块的特征.收集到的2676个腺苷酰化结构域(包括302个已知功能的标签结构域)的氨基酸序列平均一致性为36.30%,其中2358个被成功地分为174个分支(不含聚酮合酶-非核糖体多肽合成酶杂合酶中的腺苷酰化结构域).由于数目较多,本文图中仅列出具有5个以上节点的分支簇,且排除了参与短链脂肪酸合成的酰基辅酶A连接酶(http://www.researchgate.net/publication/354887574_Table S1).由此产生的网络可将腺苷酰化结构域分为多模块、双模块、单模块和杂合的非核糖体多肽合成酶.这与先前报道的系统发育分析基本一致,即真菌非核糖体多肽合成酶分为两大分支:远古起源的单/双模块和新近起源的多/双模块[24, 27-28]. ...
Cyclosporine biosynthesis in Tolypocladium inflatum benefits fungal adaptation to the environment
2
2018
... 肉座菌目虫生真菌基因组预测的2440个腺苷酰化结构域中,有1056个来自于多模块的非核糖体多肽合成酶(图1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4, 24],是发现新型活性产物的宝贵资源. ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. The nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. Boxed groups show A domains from the group of enzymes that also contain DtxS1, or superclades that are formed by closely related A domain clades such as clades #52~64 (see text for details). Sub-clades of A domains are indicated with a number-letter combination, for example 52a. NRPSs of the BGCs that contain the voucher A domains include: #27a~32a, destruxin synthetase DtxS1[9]; #32b, cyclosporine synthetase[29]; #33~39, serinocyclin synthetase[13-14]; #44~45, ACVS: N-(5-amino-5-carboxypentanoyl)-L-cysteinyl-D-valine synthetase in penicillin/cephalosporin biosynthesis; #46~51, Tex1: a peptaibol synthetase from Trichoderma virens[30]; #75~76, beauvericin and bassianolide synthetase[11-12].) ...
Identification of peptaibols from Trichoderma virens and cloning of a peptaibol synthetase
4
2002
... 肉座菌目虫生真菌基因组预测的2440个腺苷酰化结构域中,有1056个来自于多模块的非核糖体多肽合成酶(图1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4, 24],是发现新型活性产物的宝贵资源. ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. The nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. Boxed groups show A domains from the group of enzymes that also contain DtxS1, or superclades that are formed by closely related A domain clades such as clades #52~64 (see text for details). Sub-clades of A domains are indicated with a number-letter combination, for example 52a. NRPSs of the BGCs that contain the voucher A domains include: #27a~32a, destruxin synthetase DtxS1[9]; #32b, cyclosporine synthetase[29]; #33~39, serinocyclin synthetase[13-14]; #44~45, ACVS: N-(5-amino-5-carboxypentanoyl)-L-cysteinyl-D-valine synthetase in penicillin/cephalosporin biosynthesis; #46~51, Tex1: a peptaibol synthetase from Trichoderma virens[30]; #75~76, beauvericin and bassianolide synthetase[11-12].) ...
... 分支46~51对应的非核糖体多肽合成酶可能产生peptaibols类线性多肽[30],其前体单元除氨基酸外,还包括由基因簇中其他酶合成的非常规前体.这些多模块非核糖体多肽合成酶大多来自Trichotheciam和Tolypocladium属真菌[30],由8个以上CAT模块组成,有的还具有特殊模块或结构域,如T. inflatum的ctg24_orf003具有一个酮酯酰合成酶和丙二酰/酰基转移酶结构域组成的加载模块.O. australis的CDD81_5223的终端结构域为硫酯酶结构域,而不是CT.这些非核糖体多肽合成酶所在的基因簇并不保守,普遍存在转氨酶基因,可能用于合成非常规氨基酸前体. ...
... [30],由8个以上CAT模块组成,有的还具有特殊模块或结构域,如T. inflatum的ctg24_orf003具有一个酮酯酰合成酶和丙二酰/酰基转移酶结构域组成的加载模块.O. australis的CDD81_5223的终端结构域为硫酯酶结构域,而不是CT.这些非核糖体多肽合成酶所在的基因簇并不保守,普遍存在转氨酶基因,可能用于合成非常规氨基酸前体. ...
Cytotoxic and antihaptotactic beauvericin analogues from precursor-directed biosynthesis with the insect pathogen Beauveria bassiana ATCC 7159
1
2007
... 除此之外,一些分支包含产物已知的非核糖体多肽合成酶,具有相同的结构域组成,所在的基因簇也高度相似,可能产生与已知化合物结构类似的产物.其中,分支75~76包含白僵菌素/白僵菌环四肽合成酶的腺苷酰化结构域[11, 31],分支27~32和33~39则分别包含破坏素[9-10]和serinocyclin[13]合成酶的腺苷酰化结构域(图1),说明这些分支中的菌株具有合成杀虫化合物的潜力. ...
A complex ergovaline gene cluster in epichloe endophytes of grasses
4
2007
... 如图3所示,单/双模块的非核糖体多肽合成酶包括赖氨酸生物合成途径中还原α-氨基己二酸的L-氨基己二酸半醛脱氢酶(分支23)[24],参与形态发育但产物未知的ChNPS10合成酶(分支15)[40],产生聚硫二酮哌嗪的ChNPS11/ETP毒素合成酶(分支12和13)等[41],其合成的代谢物参与了真菌重要的生物过程,如性发育、生殖、分生孢子发育等,因此在基因组中也较为保守[24,32-39].其中,部分基因的生理功能已有研究,但其合成产物仍然未知. ...
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
... 分支16~20的腺苷酰化结构域来自于单模块非核糖体多肽合成酶.其中,分支16主要存在于白僵菌/冬虫夏草属真菌的基因组中,它们的编码基因与羧肽酶、双加氧酶、铁(Ⅱ)加氧酶、酰胺水解酶、磷酸酯酶和转运体蛋白等基因共同组成基因簇,其中一些基因簇还编码聚酮合酶-非核糖体多肽合成酶杂合酶、酮基还原酶、烯基还原酶、甲基转移酶和细胞色素P450蛋白.分支17中的腺苷酰化结构域主要存在于绿僵菌中,它们的基因簇含有一个寡肽转运蛋白基因,但没有修饰酶基因.分支19~20中的腺苷酰化结构域存在于大多数已测序的绿僵菌属真菌中,与LpsC或LpsB型麦角生物碱非核糖体多肽合成酶十分相似[32],其基因簇中包含萜烯合成酶,可能产生麦角酸α-羟乙基酰胺型或麦角碱类产物[43]. ...
Creating and screening Cochliobolus heterostrophus non-ribosomal peptide synthetase mutants
2
2008
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
Diverse lifestyles and strategies of plant pathogenesis encoded in the genomes of eighteen dothideomycetes fungi
2
2012
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
The siderophore biosynthetic gene SID1, but not the ferroxidase gene FET3, is required for full Fusarium graminearum virulence
2
2007
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
Ferricrocin, a siderophore involved in intra- and transcellular iron distribution in Aspergillus fumigatus
3
2009
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
... 铁载体是一种低分子量的铁螯合物,可分为胞外和胞内两种类型,因与病原真菌的致病性相关而受到广泛关注[37].铁载体合成酶具有保守的结构域,合成产物的结构和生物学功能相似,在各种营养模式的真菌中广泛分布,包括动植物病原菌和腐生菌.铁载体合成酶是多模块的非核糖体多肽合成酶,在系统发育中属于较新的分支,然而由于其序列和功能的保守性,将它们与单/双模块的非核糖体多肽合成酶一起分析.大部分肉座菌目虫生真菌的基因组中都有铁载体合成酶NPS2/SidC(分支1~4)[36]、NPS6(分支9)[39]、SidN(分支7和8)[38]和/或SidI(分支21)[37]的编码基因.值得注意的是,分支5和6的腺苷酰化结构域与NPS2/SidC的第1个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在.这表明该分支的类SidC非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能.分支10~11的双模块非核糖体多肽合成酶与人类病原菌A. fumigatus的SidE相似[42].SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
Mevalonate governs interdependency of ergosterol and siderophore biosyntheses in the fungal pathogen Aspergillus fumigatus
4
2012
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
... 铁载体是一种低分子量的铁螯合物,可分为胞外和胞内两种类型,因与病原真菌的致病性相关而受到广泛关注[37].铁载体合成酶具有保守的结构域,合成产物的结构和生物学功能相似,在各种营养模式的真菌中广泛分布,包括动植物病原菌和腐生菌.铁载体合成酶是多模块的非核糖体多肽合成酶,在系统发育中属于较新的分支,然而由于其序列和功能的保守性,将它们与单/双模块的非核糖体多肽合成酶一起分析.大部分肉座菌目虫生真菌的基因组中都有铁载体合成酶NPS2/SidC(分支1~4)[36]、NPS6(分支9)[39]、SidN(分支7和8)[38]和/或SidI(分支21)[37]的编码基因.值得注意的是,分支5和6的腺苷酰化结构域与NPS2/SidC的第1个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在.这表明该分支的类SidC非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能.分支10~11的双模块非核糖体多肽合成酶与人类病原菌A. fumigatus的SidE相似[42].SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
... [37]的编码基因.值得注意的是,分支5和6的腺苷酰化结构域与NPS2/SidC的第1个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在.这表明该分支的类SidC非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能.分支10~11的双模块非核糖体多肽合成酶与人类病原菌A. fumigatus的SidE相似[42].SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
An extracellular siderophore is required to maintain the mutualistic interaction of Epichlo? festucae with Lolium perenne
3
2013
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
... 铁载体是一种低分子量的铁螯合物,可分为胞外和胞内两种类型,因与病原真菌的致病性相关而受到广泛关注[37].铁载体合成酶具有保守的结构域,合成产物的结构和生物学功能相似,在各种营养模式的真菌中广泛分布,包括动植物病原菌和腐生菌.铁载体合成酶是多模块的非核糖体多肽合成酶,在系统发育中属于较新的分支,然而由于其序列和功能的保守性,将它们与单/双模块的非核糖体多肽合成酶一起分析.大部分肉座菌目虫生真菌的基因组中都有铁载体合成酶NPS2/SidC(分支1~4)[36]、NPS6(分支9)[39]、SidN(分支7和8)[38]和/或SidI(分支21)[37]的编码基因.值得注意的是,分支5和6的腺苷酰化结构域与NPS2/SidC的第1个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在.这表明该分支的类SidC非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能.分支10~11的双模块非核糖体多肽合成酶与人类病原菌A. fumigatus的SidE相似[42].SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
NPS6, encoding a nonribosomal peptide synthetase involved in siderophore-mediated iron metabolism, is a conserved virulence determinant of plant pathogenic ascomycetes
4
2006
... 如图3所示,单/双模块的非核糖体多肽合成酶包括赖氨酸生物合成途径中还原α-氨基己二酸的L-氨基己二酸半醛脱氢酶(分支23)[24],参与形态发育但产物未知的ChNPS10合成酶(分支15)[40],产生聚硫二酮哌嗪的ChNPS11/ETP毒素合成酶(分支12和13)等[41],其合成的代谢物参与了真菌重要的生物过程,如性发育、生殖、分生孢子发育等,因此在基因组中也较为保守[24,32-39].其中,部分基因的生理功能已有研究,但其合成产物仍然未知. ...
... [其中每个节点代表一个腺苷酰化结构域,连接线粗细与相似性成正比.节点颜色显示非核糖体多肽合成酶的类型,形状表示肉座菌目虫生真菌的种类(科),已知功能的结构域用箭头表示.绿色阴影标注产物未知的非核糖体多肽合成酶腺苷酰化结构域.括号中的数字表示腺苷酰化结构域在标签非核糖体多肽合成酶中的位置,例如(1/3)表示该结构域是一个具有3个模块的非核糖体多肽合成酶的第1个腺苷酰化结构域.AAR—L-氨基己二酸半醛脱氢酶[24];LpsC和LpsB—D-麦角酰肽合成酶亚基1和2,参与麦角生物碱的生物合成[32];ChNPS10—参与真菌形态发育[33-34];NPS2/SidC[35-36]和SidI[37]—胞内铁载体合成酶;SidN[38]和NPS6[39]:胞外铁载体合成酶] ...
... (Nodes in the network represent A domains, and the line boldness is proportional to the identity. Nodes are colored according to the domain composition of NRPS. Referenced A domains from voucher NRPSs with known products are indicated by arrowheads. The predicted products/functions of the enzymes are listed below the clades. NRPSs that contain the voucher A domains include: AAR, L-aminoadipate-semialdehyde dehydrogenase[24]; LpsC and LpsB: D-lysergyl-peptide synthetase subunit 1 and 2, respectively, involved in ergot alkaloid biosynthesis[32]; ChNPS10[33-34]: an NRPS-like protein involved in morphological development; NPS2/SidC[35-36] and SidI[37]: synthetases for intracellular siderophores; SidN[38] and NPS6[39]: synthetases for extracellular siderophores. Numbers in parentheses indicate the position of the A domain in the voucher NRPS, for example (1/3) indicates the first A domain out of the three housed in the NRPS.) ...
... 铁载体是一种低分子量的铁螯合物,可分为胞外和胞内两种类型,因与病原真菌的致病性相关而受到广泛关注[37].铁载体合成酶具有保守的结构域,合成产物的结构和生物学功能相似,在各种营养模式的真菌中广泛分布,包括动植物病原菌和腐生菌.铁载体合成酶是多模块的非核糖体多肽合成酶,在系统发育中属于较新的分支,然而由于其序列和功能的保守性,将它们与单/双模块的非核糖体多肽合成酶一起分析.大部分肉座菌目虫生真菌的基因组中都有铁载体合成酶NPS2/SidC(分支1~4)[36]、NPS6(分支9)[39]、SidN(分支7和8)[38]和/或SidI(分支21)[37]的编码基因.值得注意的是,分支5和6的腺苷酰化结构域与NPS2/SidC的第1个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在.这表明该分支的类SidC非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能.分支10~11的双模块非核糖体多肽合成酶与人类病原菌A. fumigatus的SidE相似[42].SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
The genome of Tolypocladium inflatum: Evolution, organization, and expression of the cyclosporin biosynthetic gene cluster
1
2013
... 如图3所示,单/双模块的非核糖体多肽合成酶包括赖氨酸生物合成途径中还原α-氨基己二酸的L-氨基己二酸半醛脱氢酶(分支23)[24],参与形态发育但产物未知的ChNPS10合成酶(分支15)[40],产生聚硫二酮哌嗪的ChNPS11/ETP毒素合成酶(分支12和13)等[41],其合成的代谢物参与了真菌重要的生物过程,如性发育、生殖、分生孢子发育等,因此在基因组中也较为保守[24,32-39].其中,部分基因的生理功能已有研究,但其合成产物仍然未知. ...
The epipolythiodiketopiperazine gene cluster in claviceps purpurea: Dysfunctional cytochrome P450 enzyme prevents formation of the previously unknown clapurines
1
2016
... 如图3所示,单/双模块的非核糖体多肽合成酶包括赖氨酸生物合成途径中还原α-氨基己二酸的L-氨基己二酸半醛脱氢酶(分支23)[24],参与形态发育但产物未知的ChNPS10合成酶(分支15)[40],产生聚硫二酮哌嗪的ChNPS11/ETP毒素合成酶(分支12和13)等[41],其合成的代谢物参与了真菌重要的生物过程,如性发育、生殖、分生孢子发育等,因此在基因组中也较为保守[24,32-39].其中,部分基因的生理功能已有研究,但其合成产物仍然未知. ...
Bimodular peptide synthetase SidE produces fumarylalanine in the human pathogen Aspergillus fumigatus
2
2013
... 铁载体是一种低分子量的铁螯合物,可分为胞外和胞内两种类型,因与病原真菌的致病性相关而受到广泛关注[37].铁载体合成酶具有保守的结构域,合成产物的结构和生物学功能相似,在各种营养模式的真菌中广泛分布,包括动植物病原菌和腐生菌.铁载体合成酶是多模块的非核糖体多肽合成酶,在系统发育中属于较新的分支,然而由于其序列和功能的保守性,将它们与单/双模块的非核糖体多肽合成酶一起分析.大部分肉座菌目虫生真菌的基因组中都有铁载体合成酶NPS2/SidC(分支1~4)[36]、NPS6(分支9)[39]、SidN(分支7和8)[38]和/或SidI(分支21)[37]的编码基因.值得注意的是,分支5和6的腺苷酰化结构域与NPS2/SidC的第1个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在.这表明该分支的类SidC非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能.分支10~11的双模块非核糖体多肽合成酶与人类病原菌A. fumigatus的SidE相似[42].SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
... [42].在球孢白僵菌中,类SidE编码基因BBA_07589在感染小菜蛾期间上调[27].因此,分支10~11的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击. ...
Molecular genetics of secondary chemistry in Metarhizium fungi
1
2016
... 分支16~20的腺苷酰化结构域来自于单模块非核糖体多肽合成酶.其中,分支16主要存在于白僵菌/冬虫夏草属真菌的基因组中,它们的编码基因与羧肽酶、双加氧酶、铁(Ⅱ)加氧酶、酰胺水解酶、磷酸酯酶和转运体蛋白等基因共同组成基因簇,其中一些基因簇还编码聚酮合酶-非核糖体多肽合成酶杂合酶、酮基还原酶、烯基还原酶、甲基转移酶和细胞色素P450蛋白.分支17中的腺苷酰化结构域主要存在于绿僵菌中,它们的基因簇含有一个寡肽转运蛋白基因,但没有修饰酶基因.分支19~20中的腺苷酰化结构域存在于大多数已测序的绿僵菌属真菌中,与LpsC或LpsB型麦角生物碱非核糖体多肽合成酶十分相似[32],其基因簇中包含萜烯合成酶,可能产生麦角酸α-羟乙基酰胺型或麦角碱类产物[43]. ...