|
||
|
合成基因线路的工程化设计研究进展与展望
合成生物学
2025, 6 (1):
45-64.
DOI:10.12211/2096-8280.2023-096
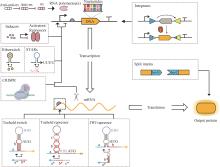
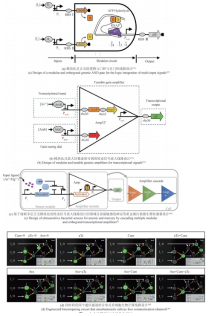
合成基因线路利用合成生物学的技术和方法,将生物元件进行重新设计与构建,使人工设计的生物分子线路在活细胞中行使特定生物功能,在生物制造、医疗健康以及环境监测等领域具有巨大的潜力。但其工程化设计仍受到各种因素的制约,包括正交元器件数量有限、大规模线路组装困难、线路行为预测性低等。根据研究者们开发的各种调控元件工具箱和组装方法,本文逐点阐述了工程化设计基因线路所需遵循的几个核心原则:正交化、标准化、模块化与自动化。文章从DNA复制、转录和翻译层面介绍了正交基因元件库的构建和改造方法;全面总结了基因元件的标准化定量表征方法与标准元件设计方法;并介绍了本团队与其他团队在模块化基因线路设计方面的相关进展;分别从软件、硬件和人工智能角度展示如何实现基因线路的自动化设计。最后,本文探讨了基因线路设计的未来发展趋势,指出需要进一步融合人工智能和自动化等信息技术来加速基因线路“设计-构建-测试-学习”循环的迭代,提高线路设计的功能可预测性和复杂性,高效设计出符合目标需求的人造生命体。  View image in article
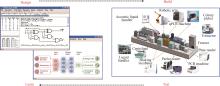
图2
基因元器件精选数据库BioPartsDB的网站结构与内容设计示意图[
(a) BioPartsDB数据库的设计架构图,箭头代表数据库里的预期用户流,箭头往下越来越多的堆叠面板代表该部分的页面数量越多,包含的信息越详细;(b)具体到某个基因元件数据的页面内容示例;(c)某种类型的基因元件列表页面内容示例,显示元件的简要说明和关键性能数据信息
正文中引用本图/表的段落
同时,在基因元件的表征方面,目前缺少标准化的评定方法,这就造成已有的如Registry for Standard Biological Parts和SynBioHub[72]基因元件存储库中的各元件之间无法进行准确的定量比较。研究者只能在同时表征大量基因元件的研究文献中获得具有可比性的数据,但归纳总结这类研究,并获得理想的数据十分低效耗时。为此,本团队对已经过实验验证和文献报道的标准基因元器件及其表征数据进行了系统的分类,建立了BioPartsDB(
本文的其它图/表
|