|
||
|
Synthetic genetic circuit engineering: principles, advances and prospects
Synthetic Biology Journal
2025, 6 (1):
45-64.
DOI: 10.12211/2096-8280.2023-096
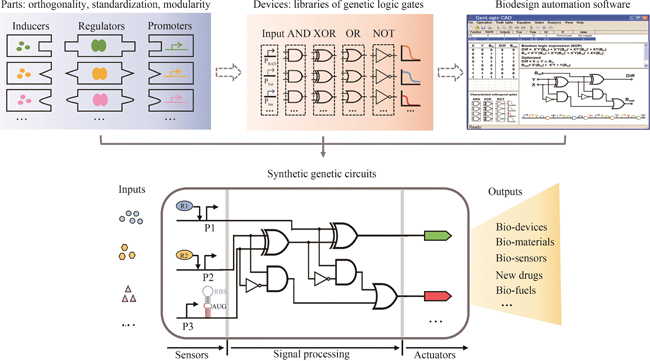
Synthetic genetic circuits are engineered gene networks comprised of redesigned genetic parts for interacting to perform customized functions in cells. With the rapid development of synthetic biology, synthetic genetic circuits have shown significant application potentials in many fields such as biomanufacturing, healthcare and environmental monitoring. However, the efforts to scale up genetic circuits are hindered by the limited number of orthogonal parts, the difficulty of functionally composing large-scale circuits, and the poor predictability of circuit behaviors. A longstanding goal of synthetic biology research is to engineer complex synthetic biological circuits, using modular genetic parts, as we do with electronic circuits. Synthetic biologists have developed various genetic toolboxes and functional assembly methods over the past few decades. Here we present an overview of the latest advances, challenges, and future prospects in genetic circuit engineering from four aspects corresponding to the four key engineering principles for circuit design, i.e. orthogonality, standardization, modularity, and automation. Firstly, the design and construction of orthogonal genetic part libraries are discussed in both prokaryotes and eukaryotes at the levels of DNA replication, transcription, and translation, respectively. Standardized characterization methods and the design of modular genetic parts are subsequently summarized. Furthermore, progress in developing modular genetic circuits are presented, providing new concepts and ways for engineering increasingly large and complex circuits. Finally, how to achieve automated design and building of genetic circuits are addressed from the advances in software, hardware and artificial intelligence, respectively, with an aim to replacing the presently time-consuming manual trial-and-error mode with the iterative "design-build-test-learn" cycle for improved efficiency and predictability of circuit design. The integration of these fundamental principles and the latest advances in information technology such as artificial intelligence and lab automation will accelerate the paradigm shift in genetic circuit engineering and synthetic biology research, making it feasible for designing synthetic lives to meet various customized needs.
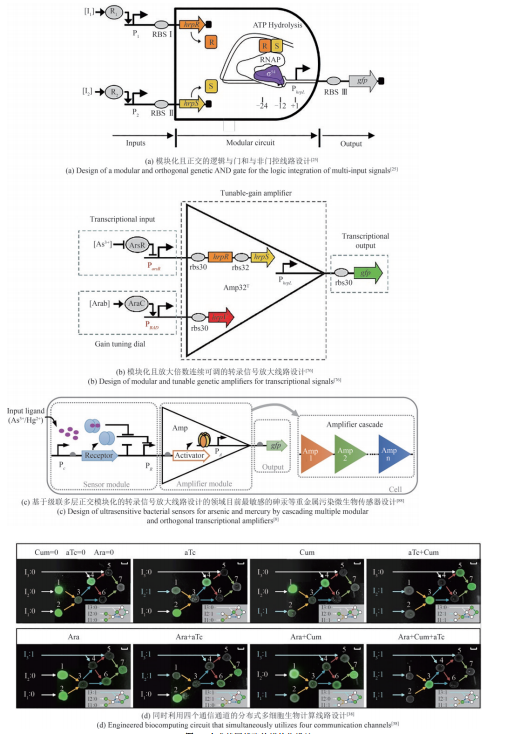
Fig. 3
Modular design of synthetic genetic circuits
Extracts from the Article
模块化、不受细胞类型影响且可编程的系统是合成生物学研究中的重要瓶颈问题,相关的研究不断在扩充模块化的基因线路,使其不受转录、翻译资源影响,具备可预测性。本团队在标准化工作环境下充分测试基因线路各组成部件特性,利用铜绿假单胞菌中的异源σ54依赖型转录因子和相关基因调控元件在大肠杆菌中率先设计并构建了模块化且正交化的逻辑与门和与非门控线路、模块化且放大倍数连续可调的转录信号放大线路[图3(a)和(b)][25, 76]。并通过结合现有的sgRNA支架设计和一种已被证明模块性较高的σ54因子依赖型激活子PspF,在模式生物大肠杆菌(Escherichia coli)和固氮菌(Klebsiella oxytoca)中实现了类真核CRISPR激活,并且激活装置具有泄露表达水平低、激活倍率高等优异的调控性能[45]。经过进一步的工程化改造和优化,其基因调控性能第一次达到了足以构建CRISPRa级联调控、CRISPRa正反馈调控和多通道基因调控的水平上。Li等[77]在哺乳动物细胞中结合新设计的杂交启动子并通过关键转录起始元件的空间位阻发挥转录抑制作用,构建了包含26种可逆转录激活因子样效应抑制因子(transcription activator-like effector repressors,TALER)的文库,可以准确预测模块化组装的TALER级联和开关电路的行为。很多天然的转录因子已经被深入研究并应用于代谢通路动态调控和人工基因线路构建[78]。然而,天然的转录因子的调控性能难以满足工程化设计的需求,传统筛选和定向进化方法可改善转录因子的性能,但相关实验周期长、工作量大。Hou等[79]针对转录因子的协同性、超敏性需求,开发了一种模块化的协同性蛋白元件,将该元件与转录因子融合后,可有效提高转录因子结合DNA的协同性,从而显著提升转录调控系统的超敏性和严谨性。该元件普遍适用于各种转录因子,在不同物种中也具有很高的通用性,可对遗传开关和人工调控网络实行“即插即用”式的改造,大幅改善人工系统的性能。
复杂基因线路中基因模块的行为通常是不可预测的[80]。在组装模块为基因线路后,下游元件(例如调节蛋白的结合位点)会对上游模块产生负载,从而对预测的行为产生干扰[81-83]。Ceroni等[84]开发了一套用单位细胞产生的荧光量反映人工基因元件对细胞资源的占用情况的检测方法,该方法首次建立模型来计算可使用的细胞资源,有助于选择出最优的基因线路设计方案。细胞生长的内部环境是动态变化的,这使得构建基因线路或者网络时,在细胞中引入的元件难以实现稳定的输出,如众所熟知的启动子。MIT Voigt实验室的研究人员运用控制论理论设计了能够在任何拷贝数下实现目的基因同一水平表达的启动子元件,研究人员利用transcription-activator-like effector(TALE)构建了iFFL稳定的启动子(iFFLs-stabilized promoter)。结果表明,即使基因的拷贝数由于基因组突变或者培养基条件改变发生了变化,这些iFFL稳定的启动子控制的目的基因在不同的基因组位点或者质粒上仍能实现接近相同的表达水平[85]。本团队首次证明了人工合成核酸海绵可系统地调节基因线路中的基因表达,从而精确改变该线路中的基因表达泄露、输出幅度和诱导倍数、对小分子的响应灵敏度,并改善宿主细胞的生长速度[86]。该基因调节方法简单、有效,可广泛用于多种应用领域相关的人工合成基因线路设计。Mishra等[87]设计了一种包含快速磷酸转移过程的负载驱动器,即使在存在大量负载的情况下也能响应时变输入信号,并且在酿酒酵母中测试了该模块的有效性。该负载驱动器可作为创建复杂的、更高级别的基因线路的基本构建块。本团队通过级联多层正交模块化的转录信号放大线路,首先通过调节细胞内受体蛋白的密度来提高传感器的灵敏度,其次通过串联多极转录放大器来促进输出模块的信号强度,最后结合转录调节和翻译后调节的方法来降低在无输入信号时传感器的信号输出,从而增加输出信号的动态范围,创建了领域目前最敏感的基于大肠杆菌的砷和汞等重金属污染细胞传感器及创新的可视化输出模块[图3(c)][88]。Liu等[89]揭示了Ⅱ型CRISPR系统crRNA-tracrRNA配对的可编程性机制,利用重编程的tracrRNA劫持各种RNA分子激活CRISPR系统,并利用双gRNA介导的CRISPR激活(CRISPRa)系统,构建了一系列正交化的可编程AND-gate布尔逻辑门库,为编程生物学带来了更多可能性,包括重组细胞内的基因调控网络拓扑结构、开发新的RNA编辑器和RNA标记方法、设计复杂细胞生物计算线路等。
同时,单个细胞的负载有限,组合多种功能细胞,可以提高群体对复杂环境的适应性和鲁棒性,以完成更加复杂的工作。可通过多细胞分布式线路设计,让单个细胞里的线路模块化并进行多个细胞间的级联通信来提高线路的模块性和实现线路的规模化组装设计。相比于基于单种细胞的计算线路,分布式计算不仅可以降低每个细胞中合成线路的体积,减少代谢负担,而且可以利用细胞高度模块化的特性,在不同的细胞群体内重复使用相同的基因元件,降低对正交基因调控元件的依赖。为了演示更复杂的多细胞生物计算功能,研究人员设计了复杂的三输入XOR-AND逻辑门电路。XOR-AND逻辑门电路分别部署在七个不同的大肠杆菌菌株中,由四个通信通道协调。每个菌株包含一个NOR门(cell-1至cell-6)或一个Buffer门(cell-7),这是已知第一个同时利用四个通信通道的生物计算线路[图3(d)][38]。虽然多细胞分布式计算在细菌、酵母和哺乳动物细胞中都已经实现,但它们的规模却并未得到显著的提高。在细菌中最具有代表性的分布式计算线路仍然是基于多细胞或非门的16个2输入逻辑门以及更复杂一些的与门-异或门复合逻辑[3, 38]。而在酵母与哺乳动物细胞中,分布式计算线路的规模也与单细胞计算线路的规模相差不大,最具代表性的是1位全加器程序[90-91]。
Other Images/Table from this Article
|