|
||
|
Applications of the CRISPR/Cas9 editing system in the study of microbial natural products
Synthetic Biology Journal
2024, 5 (3):
658-671.
DOI: 10.12211/2096-8280.2023-110
Microorganisms have consistently been a crucial source for researchers to explore and develop new natural products. Currently, research methods involving gene editing tools for the discovery, biosynthesis, and metabolic engineering of natural products have garnered broad attention in this field. However, traditional methods for gene editing usually rely on the recombination ability of the host or introduced proteins. It’s difficult to establish a general platform for all bacteria mainly because of their complicated genetic background. This genetic diversity often causes laborious experimental operations with low efficiency. The CRISPR/Cas9 gene editing system, with its unique and flexible targeting advantages, overcomes common limitations such as sequence homology or site constraint in other gene editing methods and thus is more likely to function in diverse bacteria species. This simplifies experimental procedures, enhances work efficiency, and promotes the development of natural product research. This article introduces the applications of the CRISPR/Cas9 system for the discovery, biosynthesis, and metabolic engineering of natural products in microorganisms. It covers the development of the CRISPR/Cas9 system, cloning and genetic editing of natural product biosynthetic gene clusters, structural derivatization and metabolic engineering of natural products, and the activation of silenced natural product biosynthetic gene clusters. These aspects highlight the advantages of the CRISPR/Cas9 system in the research of natural products with microorganisms. Finally, solutions are proposed for addressing challenges that the CRISPR/Cas9 system currently faces in overcoming low recombination efficiency and host adaptability issues. Especially the CRISPR/Cas12a system which has broadened applications of the CRISRP/Cas9 system by preferring different PAM sites. In addition to functions that CRISPR/Cas9 system has realized, its potent multiple targeting ability further enhances the efficiency of target editing. It is believed that with the development of synthetic biology and information technology, an increasing number of genetic manipulation tools and methods related to the CRISPR/Cas9 system will be developed, continually driving progress in the research of natural products.
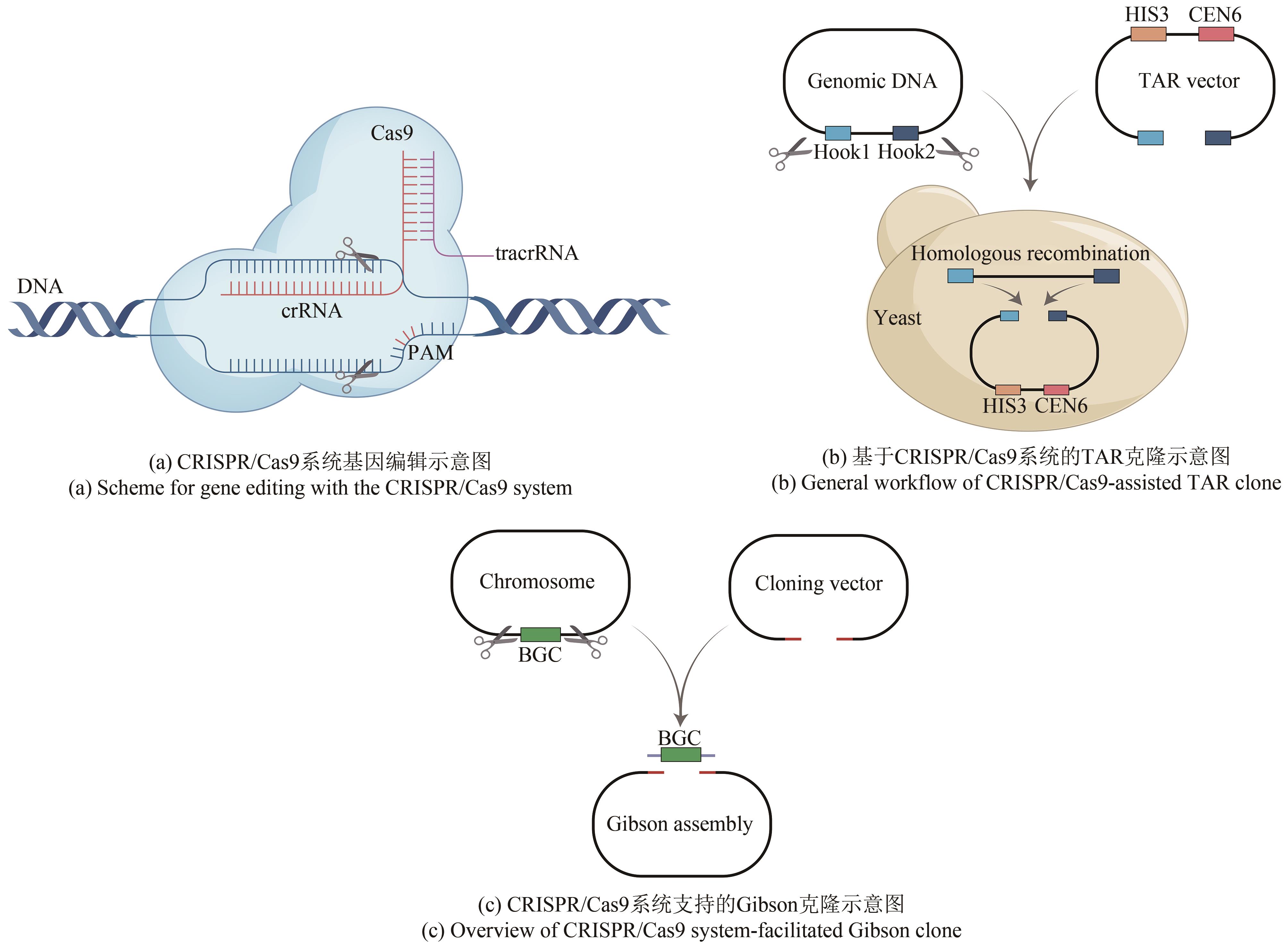
Fig. 1
CRISPR/Cas9-assisted directly cloning of biosynthetic gene clusters
Extracts from the Article
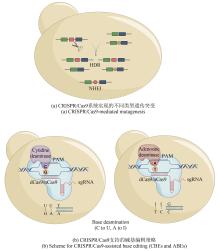
随后,在众多研究团队的努力下,研究者揭示并阐明了该系统的最小基本单元组成、靶点特异性决定因素以及加工剪切过程[图1(a)]。简而言之,由CRISPR loci转录得到的pre-crRNA,在部分互补的tracrRNA(trans-encoded small RNA)、宿主RNaseⅢ以及Cas9蛋白的帮助下完成gRNA的成熟。然后,Cas9蛋白和gRNA组成的复合物对外源DNA进行匹配,Cas9在特异性识别PAM(protospacer-adjacent motif)位点后解开DNA双链,进行gRNA互补配对。最后,在正确配对情况下,Cas9蛋白通过其HNH和RuvC核酸内切酶结构域在非互补链的PAM位点上游3 bp处切割DNA双链[10-11]。CRISPR/Cas9系统高效且特异性的双链切割能力促使研究人员开发了多种灵活多样的基因编辑工具,通过质粒表达CRISPR/Cas9系统实现体内双链剪切,或通过Cas9蛋白和gRNA实现体外靶向位点切割。其中以借助CRISPR/Cas9系统的靶向切割实现同源重组菌株的高效筛选被广泛使用[12-15]。
Richard F. Lockey团队和Natalay Kouprina团队分别利用CRISPR/Cas9系统辅助了Gibson组装和TAR克隆。前者通过Cas9和gRNA的体外反应实现质粒的靶向切割,随后和一个末端同切割后质粒同源的目的序列退火连接,最终实现Gibson组装[53]。后者则利用体内对目的基因组的靶向切割,借助酵母的高效同源重组能力,从而实现了靶向基因序列的高效TAR克隆[54][图1(b)]。这些研究为利用CRISPR/Cas9系统实现任意目的基因序列的大片段克隆提供了可能。清华大学的朱听团队[29]开发了CATCH(Cas9-assisted targeting of chromosome segments)系统靶向大的生物合成基因簇[图1(c)]。借助靶向DNA序列末端同源的线性化质粒,通过gRNA实现Cas9蛋白对靶向DNA序列特异性切割从而暴露出和线性化质粒末端同源的序列,最终实现Gibson组装。该团队同时测试了来自不同菌株的不同长度片段,高阳性率证实了该捕获技术可实现50 kb到150 kb序列的克隆。此外,Olga Genilloud团队[30]借助CATCH技术从Streptomyces cacaoi CA-170360菌株中成功地克隆了cpp基因簇,并验证了该串联基因簇是两个环肽的生物合成来源。
Richard F. Lockey团队和Natalay Kouprina团队分别利用CRISPR/Cas9系统辅助了Gibson组装和TAR克隆.前者通过Cas9和gRNA的体外反应实现质粒的靶向切割,随后和一个末端同切割后质粒同源的目的序列退火连接,最终实现Gibson组装[
Richard F. Lockey团队和Natalay Kouprina团队分别利用CRISPR/Cas9系统辅助了Gibson组装和TAR克隆.前者通过Cas9和gRNA的体外反应实现质粒的靶向切割,随后和一个末端同切割后质粒同源的目的序列退火连接,最终实现Gibson组装[
Richard F. Lockey团队和Natalay Kouprina团队分别利用CRISPR/Cas9系统辅助了Gibson组装和TAR克隆.前者通过Cas9和gRNA的体外反应实现质粒的靶向切割,随后和一个末端同切割后质粒同源的目的序列退火连接,最终实现Gibson组装[
Richard F. Lockey团队和Natalay Kouprina团队分别利用CRISPR/Cas9系统辅助了Gibson组装和TAR克隆.前者通过Cas9和gRNA的体外反应实现质粒的靶向切割,随后和一个末端同切割后质粒同源的目的序列退火连接,最终实现Gibson组装[
Richard F. Lockey团队和Natalay Kouprina团队分别利用CRISPR/Cas9系统辅助了Gibson组装和TAR克隆.前者通过Cas9和gRNA的体外反应实现质粒的靶向切割,随后和一个末端同切割后质粒同源的目的序列退火连接,最终实现Gibson组装[
Other Images/Table from this Article
|