Biofuels: environment, technology and food security
1
2009
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Barriers and opportunities in bio-based production of hydrocarbons
1
2018
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
C1-based biomanufacturing: advances, challenges and perspectives
1
2023
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Microbial utilization of next-generation feedstocks for the biomanufacturing of value-added chemicals and food ingredients
1
2022
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Methylotrophs: from C1 compounds to food
1
2022
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Production of the biocommodities butanol and acetone from methanol with fluorescent FAST-tagged proteins using metabolically engineered strains of Eubacterium limosum
1
2021
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Conversion of carbon dioxide into fuels—a review
1
2022
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Metabolic engineering of Methylorubrum extorquens AM1 for poly (3-hydroxybutyrate-co-3-hydroxyvalerate) production using formate
1
2021
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Recent trends of biotechnological production of polyhydroxyalkanoates from C1 carbon sources
1
2023
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Improved methanol-derived lovastatin production through enhancement of the biosynthetic pathway and intracellular lovastatin efflux in methylotrophic yeast
1
2018
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Engineered monoculture and co-culture of methylotrophic yeast for de novo production of monacolin J and lovastatin from methanol
1
2018
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Microbial synthesis gas utilization and ways to resolve kinetic and mass-transfer limitations
1
2015
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Renewable methanol and formate as microbial feedstocks
1
2020
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Metabolic engineering strategies to enable microbial utilization of C1 feedstocks
1
2021
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Matching emerging formic acid synthesis processes with application requirements
1
2022
... 传统化工行业依赖于化石燃料或糖类资源,在实际生产过程中不仅产生了大量温室气体,对环境造成了巨大压力,而且还会与食品行业竞争,影响粮食安全[1-2].近年来,随着微生物细胞工厂的开发与应用,二氧化碳(CO2)、一氧化碳(CO)、甲烷(CH4)、甲醇与甲酸等一碳资源的生物炼制备受关注.相比于化石燃料与传统碳源,一碳资源天然储备丰富且生产成本相对低廉,因此,一碳资源有望成为下一代原料用于制备高附加值化学品[3-5],如生物能源[6-7]、生物基材料[8-9]、生物医药[10-11]等,从而推动碳资源的可持续循环利用.CO2、CO与CH4作为气态底物,在气液传质上具有一定的局限性[4,12],不利于微生物的细胞生长与产物合成;甲醇与甲酸作为液态底物,很好地绕过了这些限制条件[13].甲醇作为一种大宗化学品,产能丰富且生产成本低,但是受到易燃性的限制,甲醇在微生物培养过程中的安全性明显低于甲酸[14].因此,甲酸的工业应用逐步展现出了巨大的潜能[15],从而凸显了甲酸生物经济的优势. ...
Replacing the Calvin cycle with the reductive glycine pathway in Cupriavidus necator
5
2020
... 甲酸生物经济的发展关键在于甲酸营养型微生物的构建与应用.目前,甲酸营养型微生物的构建方式主要有两种:①优化天然甲酸利用微生物,提高甲酸代谢能力.例如,在钩虫贪铜菌(Cupriavidus necator)中,采用低能耗的异源还原性甘氨酸途径取代高能耗的本源卡尔文循环,有效提高了菌株的甲酸代谢潜力[16].虽然天然甲酸利用微生物具有一定的甲酸代谢基础,能够进行代谢工程改造,但是天然甲酸利用微生物的分子遗传学操作工具与方法相对较少,从而在很大程度上限制了其改造与应用的空间.②构建人工甲酸利用微生物,人工赋予模式微生物甲酸代谢能力.例如,通过在大肠杆菌(Escherichia coli)中引入还原性甘氨酸途径与能量再生模块,使得工程菌株能够利用甲酸为唯一碳源和能源进行细胞生长[17-18].类似地,通过在酿酒酵母(Saccharomyces cerevisiae)中强化还原性甘氨酸途径,获得的工程菌株可以同化利用甲酸与CO2[19].相比于天然甲酸利用微生物,模式微生物的代谢工程改造难度小且应用范围广,但是却面临甲酸代谢能力不足与生长速率偏低等问题,因此,需要采用合适的代谢工程策略进行相应的改造与优化,提高甲酸的利用效率. ...
... Fdh(内源酶)
[16] | | Clostridium pasteurianum | GCS(源自Gottschalkia acidurici) ...
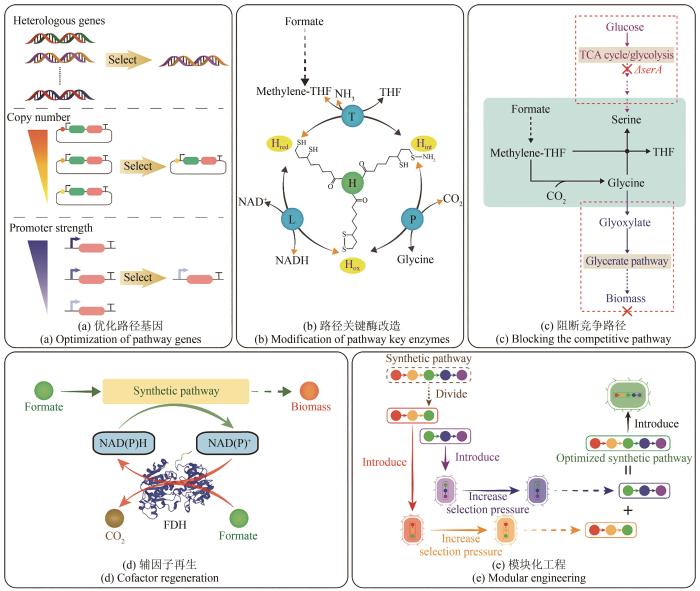
... 甲酸同化路径的构建方式主要包括两种:一种是将异源路径基因与微生物的内源基因进行理性组合;另一种是仅对微生物内源基因进行系统优化.然而,上述两种构建方式均会影响微生物的本源代谢网络,从而影响甲酸同化效率.因此,需要对路径基因的表达水平进行合理优化[图6(a)].采用的策略主要包括两种:①选择合适的异源基因,用于构建甲酸同化路径.例如,当在E. coli中引入甲酸四氢叶酸连接酶(Ftl)、次甲基四氢叶酸环水解酶(Fch)和亚甲基四氢叶酸脱氢酶(Mtd)等基因构建THF循环[图4(d)]时,同时将源自C. ljungdahlii和A. woodie的基因操纵子Cl和Aw分别引入丝氨酸营养缺陷型菌株中,结果显示Cl操纵子可以回补菌株的丝氨酸营养缺陷,使其以甲酸为碳源进行生长,但是Aw操纵子却不能实现上述效果.上述结果表明,Cl操纵子有利于异源Ftl、Fch和Mtd以及内源丝氨酸羟甲基转移酶(GlyA)的催化作用,实现甲酸到丝氨酸的转化[61].然而,上述研究所构建的THF循环对甲酸的同化效率明显低于源自M. extorquens的THF循环[62-63],这也进一步证明了选择合适的异源基因对于构建甲酸同化路径的重要性.②优化路径基因水平,实现路径与宿主的最优适配.例如,采用高拷贝数、中拷贝数和低拷贝数的质粒,在E. coli中表达Ftl、Fch、Mtd与两种甲酸脱氢酶,结果显示含有低拷贝数质粒的菌株生长速率明显高于中含有高拷贝数的菌株.上述结果表明,低拷贝数质粒有利于实现菌株路径基因的最优表达[18].除了利用不同拷贝数的质粒外,启动子工程也可以用于优化路径基因表达水平.例如,通过将不同启动子组合的质粒pC1(表达Ftl、Fch、Mtd)和pC2(表达GCS)分别导入E. coli甘氨酸营养缺陷型菌株,结果显示当采用弱启动子时菌株生长最好.上述结果表明,弱启动子组合有利于实现路径基因的最优表达[16].另外,值得注意的是,虽然使用质粒过表达路径基因相对简捷,但是过多质粒的使用会影响微生物的生长,相比而言基因组的过表达更加稳定.例如,首先在E. coli的基因组上过表达GCS,随后导入rTHF循环,获得了菌株E. coli RG3.与使用质粒过表达GCS的菌株E. coli RG2相比,E. coli RG3具有更高的生长速率与产物产率[63]. ...
... 由于甲酸同化路径与微生物的本源代谢网络存在一定的交叉性,所以不可避免地存在着路径代谢流的泄露,因此,需要对甲酸同化路径的竞争路径进行合理阻断.以rTHF-rgcv途径为例[图6(c)],一方面阻断本源代谢网络中能够合成甲酸同化途径中间产物的路径.例如,通过在serA基因(编码3-磷酸甘油酸脱氢酶)缺陷型E. coli中构建rTHF-rgcv途径,促进5,10-亚甲基四氢叶酸的合成,使得rTHF-rgcv途径的丙酮酸合成通量达到了总丙酮酸合成通量的12.9%,而在未敲除serA基因的菌株中仅为7.3%[63].另一方面阻断能够代谢甲酸同化途径中间产物的路径.例如,C. necator能够通过乙醛酸路径同化甘氨酸,即甘氨酸先氧化生成乙醛酸,随后乙醛酸通过甘油酸途径转化为生物量.然而,相比于rTHF-rgcv途径中的丝氨酸路径,乙醛酸路径同化甘氨酸的效率更低,故丝氨酸路径取代乙醛酸路径更有利于菌株在甲酸上生长[16]. ...
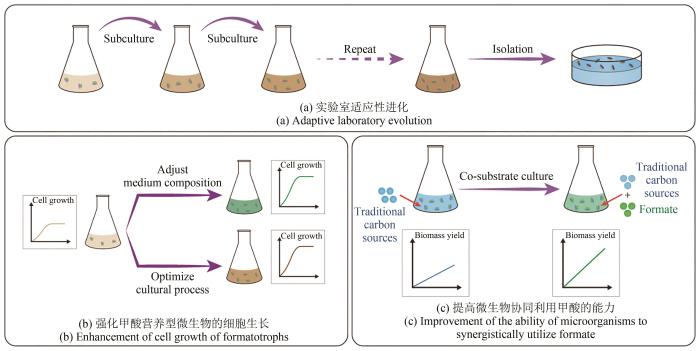
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Growth of E. coli on formate and methanol via the reductive glycine pathway
8
2020
... 甲酸生物经济的发展关键在于甲酸营养型微生物的构建与应用.目前,甲酸营养型微生物的构建方式主要有两种:①优化天然甲酸利用微生物,提高甲酸代谢能力.例如,在钩虫贪铜菌(Cupriavidus necator)中,采用低能耗的异源还原性甘氨酸途径取代高能耗的本源卡尔文循环,有效提高了菌株的甲酸代谢潜力[16].虽然天然甲酸利用微生物具有一定的甲酸代谢基础,能够进行代谢工程改造,但是天然甲酸利用微生物的分子遗传学操作工具与方法相对较少,从而在很大程度上限制了其改造与应用的空间.②构建人工甲酸利用微生物,人工赋予模式微生物甲酸代谢能力.例如,通过在大肠杆菌(Escherichia coli)中引入还原性甘氨酸途径与能量再生模块,使得工程菌株能够利用甲酸为唯一碳源和能源进行细胞生长[17-18].类似地,通过在酿酒酵母(Saccharomyces cerevisiae)中强化还原性甘氨酸途径,获得的工程菌株可以同化利用甲酸与CO2[19].相比于天然甲酸利用微生物,模式微生物的代谢工程改造难度小且应用范围广,但是却面临甲酸代谢能力不足与生长速率偏低等问题,因此,需要采用合适的代谢工程策略进行相应的改造与优化,提高甲酸的利用效率. ...
... 通过实验室适应性进化与培养条件优化等策略提高菌株对甲酸的耐受性 | [17-18,56-67] |
| 酿酒酵母 | rTHF-rgcv途径 的关键模块 | 遗传背景清晰; ...
... Fdh(源自Pseudomonas sp.) | [17] |
Escherichia coli ...
... (Kim等[17]研究的进一步拓展) ...
... 路径模块化优化作为一种理性的代谢工程策略,通过将甲酸同化路径划分为几个代谢模块,并利用相应的营养缺陷型菌株测试各个模块的活性与限制性瓶颈,通过解除瓶颈最终实现甲酸同化路径的整体优化与适配[96][图6(e)].例如,借助模块化工程策略,通过组合rTHF模块与丝氨酸-苏氨酸循环,验证了甲酸同化在E. coli中的代谢可行性[60].首先,通过在一碳与甘氨酸营养缺陷型菌株中过表达异源Ftl,甲酸能够回补菌株的一碳营养缺陷,从而证明了一碳模块的活性;其次,通过在丝氨酸营养缺陷型菌株中过表达rTHF模块的Fch/ Mtd双功能酶(FolD)与GlyA,工程菌株实现了在甲酸与甘氨酸上进行生长;最后,通过在丝氨酸营养缺陷型菌株中引入丝氨酸-苏氨酸循环,使工程菌株能够在甲酸与葡萄糖上进行细胞生长,不再需要提供外源甘氨酸.上述研究为后续甲酸同化路径的构建提供了重要参考,模块化工程不仅实现了甲酸营养型E. coli的构建[17],而且还完成了P. putida甲酸代谢改造[70]等,从而拓展了同化甲酸的底盘微生物. ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
... [17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
... 对于人工构建的甲酸营养型菌株而言,合适的培养条件对于提高菌株对甲酸的耐受性与利用效率至关重要.甲酸营养型微生物的培养条件优化主要包括两种方式[图7(b)].①培养基成分的调整.例如,基于以甲酸为唯一碳源和能源进行生长的E. coli工程菌株,通过在培养基中添加100 mmol/L的碳酸氢钠,可以使菌株在更高的甲酸浓度下进行生长,甚至可以耐受300 mmol/L甲酸.菌株对甲酸耐受性的增加可能是由于含有碳酸氢盐的溶液具有更好的pH缓冲性能,从而减少了由于甲酸消耗所引起的生长环境局部pH波动[17].②培养过程的优化.例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,并降低菌株的培养温度(由37 ℃降至32 ℃),提高了菌株细胞色素Cyo的表达水平并降低了Cyd的表达水平,使得还原力NAD(P)H能够更有效地转化为ATP,从而解决了菌株生长过程中能量供应不足的问题[18]. ...
Escherichia coli is engineered to grow on CO2 and formic acid
6
2020
... 甲酸生物经济的发展关键在于甲酸营养型微生物的构建与应用.目前,甲酸营养型微生物的构建方式主要有两种:①优化天然甲酸利用微生物,提高甲酸代谢能力.例如,在钩虫贪铜菌(Cupriavidus necator)中,采用低能耗的异源还原性甘氨酸途径取代高能耗的本源卡尔文循环,有效提高了菌株的甲酸代谢潜力[16].虽然天然甲酸利用微生物具有一定的甲酸代谢基础,能够进行代谢工程改造,但是天然甲酸利用微生物的分子遗传学操作工具与方法相对较少,从而在很大程度上限制了其改造与应用的空间.②构建人工甲酸利用微生物,人工赋予模式微生物甲酸代谢能力.例如,通过在大肠杆菌(Escherichia coli)中引入还原性甘氨酸途径与能量再生模块,使得工程菌株能够利用甲酸为唯一碳源和能源进行细胞生长[17-18].类似地,通过在酿酒酵母(Saccharomyces cerevisiae)中强化还原性甘氨酸途径,获得的工程菌株可以同化利用甲酸与CO2[19].相比于天然甲酸利用微生物,模式微生物的代谢工程改造难度小且应用范围广,但是却面临甲酸代谢能力不足与生长速率偏低等问题,因此,需要采用合适的代谢工程策略进行相应的改造与优化,提高甲酸的利用效率. ...
... 通过实验室适应性进化与培养条件优化等策略提高菌株对甲酸的耐受性 | [17-18,56-67] |
| 酿酒酵母 | rTHF-rgcv途径 的关键模块 | 遗传背景清晰; ...
... Fdh(源自Candida boidinii、Arabidopsis thaliana) | [18] |
Escherichia coli ...
... 甲酸同化路径的构建方式主要包括两种:一种是将异源路径基因与微生物的内源基因进行理性组合;另一种是仅对微生物内源基因进行系统优化.然而,上述两种构建方式均会影响微生物的本源代谢网络,从而影响甲酸同化效率.因此,需要对路径基因的表达水平进行合理优化[图6(a)].采用的策略主要包括两种:①选择合适的异源基因,用于构建甲酸同化路径.例如,当在E. coli中引入甲酸四氢叶酸连接酶(Ftl)、次甲基四氢叶酸环水解酶(Fch)和亚甲基四氢叶酸脱氢酶(Mtd)等基因构建THF循环[图4(d)]时,同时将源自C. ljungdahlii和A. woodie的基因操纵子Cl和Aw分别引入丝氨酸营养缺陷型菌株中,结果显示Cl操纵子可以回补菌株的丝氨酸营养缺陷,使其以甲酸为碳源进行生长,但是Aw操纵子却不能实现上述效果.上述结果表明,Cl操纵子有利于异源Ftl、Fch和Mtd以及内源丝氨酸羟甲基转移酶(GlyA)的催化作用,实现甲酸到丝氨酸的转化[61].然而,上述研究所构建的THF循环对甲酸的同化效率明显低于源自M. extorquens的THF循环[62-63],这也进一步证明了选择合适的异源基因对于构建甲酸同化路径的重要性.②优化路径基因水平,实现路径与宿主的最优适配.例如,采用高拷贝数、中拷贝数和低拷贝数的质粒,在E. coli中表达Ftl、Fch、Mtd与两种甲酸脱氢酶,结果显示含有低拷贝数质粒的菌株生长速率明显高于中含有高拷贝数的菌株.上述结果表明,低拷贝数质粒有利于实现菌株路径基因的最优表达[18].除了利用不同拷贝数的质粒外,启动子工程也可以用于优化路径基因表达水平.例如,通过将不同启动子组合的质粒pC1(表达Ftl、Fch、Mtd)和pC2(表达GCS)分别导入E. coli甘氨酸营养缺陷型菌株,结果显示当采用弱启动子时菌株生长最好.上述结果表明,弱启动子组合有利于实现路径基因的最优表达[16].另外,值得注意的是,虽然使用质粒过表达路径基因相对简捷,但是过多质粒的使用会影响微生物的生长,相比而言基因组的过表达更加稳定.例如,首先在E. coli的基因组上过表达GCS,随后导入rTHF循环,获得了菌株E. coli RG3.与使用质粒过表达GCS的菌株E. coli RG2相比,E. coli RG3具有更高的生长速率与产物产率[63]. ...
... 辅因子NAD(P)H可以为甲酸同化路径提供还原力,而甲酸能够在甲酸脱氢酶(Fdh)的催化下将NAD(P)+转化为NAD(P)H,从而无需添加其他底物即可完成还原力供应[图6(d)].因此,辅因子再生系统重构不仅有利于提高甲酸同化路径的催化效率,而且对于甲酸营养型微生物的还原力和能量供给至关重要.例如,通过在E. coli中异源引入能够再生NADH的Fdh(来自博伊丁假丝酵母Candida boidinii)和能够再生NADPH的Fdhmut(来自拟南芥Arabidopsis thaliana),构建了能够将NAD(P)+转化为NAD(P)H的能量再生模块,结合rTHF-rgcv途径,使得工程菌株能够利用甲酸为唯一碳源和能源进行生长[18]. ...
... 对于人工构建的甲酸营养型菌株而言,合适的培养条件对于提高菌株对甲酸的耐受性与利用效率至关重要.甲酸营养型微生物的培养条件优化主要包括两种方式[图7(b)].①培养基成分的调整.例如,基于以甲酸为唯一碳源和能源进行生长的E. coli工程菌株,通过在培养基中添加100 mmol/L的碳酸氢钠,可以使菌株在更高的甲酸浓度下进行生长,甚至可以耐受300 mmol/L甲酸.菌株对甲酸耐受性的增加可能是由于含有碳酸氢盐的溶液具有更好的pH缓冲性能,从而减少了由于甲酸消耗所引起的生长环境局部pH波动[17].②培养过程的优化.例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,并降低菌株的培养温度(由37 ℃降至32 ℃),提高了菌株细胞色素Cyo的表达水平并降低了Cyd的表达水平,使得还原力NAD(P)H能够更有效地转化为ATP,从而解决了菌株生长过程中能量供应不足的问题[18]. ...
Core catalysis of the reductive glycine pathway demonstrated in yeast
4
2019
... 甲酸生物经济的发展关键在于甲酸营养型微生物的构建与应用.目前,甲酸营养型微生物的构建方式主要有两种:①优化天然甲酸利用微生物,提高甲酸代谢能力.例如,在钩虫贪铜菌(Cupriavidus necator)中,采用低能耗的异源还原性甘氨酸途径取代高能耗的本源卡尔文循环,有效提高了菌株的甲酸代谢潜力[16].虽然天然甲酸利用微生物具有一定的甲酸代谢基础,能够进行代谢工程改造,但是天然甲酸利用微生物的分子遗传学操作工具与方法相对较少,从而在很大程度上限制了其改造与应用的空间.②构建人工甲酸利用微生物,人工赋予模式微生物甲酸代谢能力.例如,通过在大肠杆菌(Escherichia coli)中引入还原性甘氨酸途径与能量再生模块,使得工程菌株能够利用甲酸为唯一碳源和能源进行细胞生长[17-18].类似地,通过在酿酒酵母(Saccharomyces cerevisiae)中强化还原性甘氨酸途径,获得的工程菌株可以同化利用甲酸与CO2[19].相比于天然甲酸利用微生物,模式微生物的代谢工程改造难度小且应用范围广,但是却面临甲酸代谢能力不足与生长速率偏低等问题,因此,需要采用合适的代谢工程策略进行相应的改造与优化,提高甲酸的利用效率. ...
... 具有内源性甲酸脱氢酶,对甲酸的耐受性较高 | 通过代谢改造进一步提高rTHF-rgcv途径对甲酸的利用效率,开发以甲酸为唯一碳源和能源的工程菌株 | [19] |
| 毕赤酵母 | 以甲酸作为P AOX1 的诱导物 | 能够利用甲醇作为唯一碳源和能源进行细胞生长; ...
... 相比于E. coli,S. cerevisiae作为真核模式微生物,虽然生长周期较长,但是其拥有高效的内源性NAD+依赖型甲酸脱氢酶,因此对甲酸的耐受性更高.此外,已有研究表明S. cerevisiae中存在还原性甘氨酸途径所需的酶,将该途径所涉及的酶进行强化表达之后,能够实现S. cerevisiae同化利用甲酸与CO2[图2(b)],并且在1~500 mmol/L甲酸范围内生长速率均能保持恒定[19],这表明S. cerevisiae具有成为高效利用甲酸宿主的潜力. ...
... Fdh(内源酶) | [19] |
| Cupriavidus necator | Ftl、Fch、Mtd(源自Methylobacterium extorquens AM1) ...
Acetogenic prokaryotes
3
2013
... 劣势:在厌氧条件下生长,不利于工业应用;代谢研究尚不完善,代谢改造工具少 | [20-22] |
| 产甲烷菌 | 专性厌氧 | 氢营养型海沼甲烷球菌 | 还原性乙酰辅酶A途径 | 优势:能够天然利用甲酸和CO2合成甲烷,在清洁能源产业方面具有应用潜能 ...
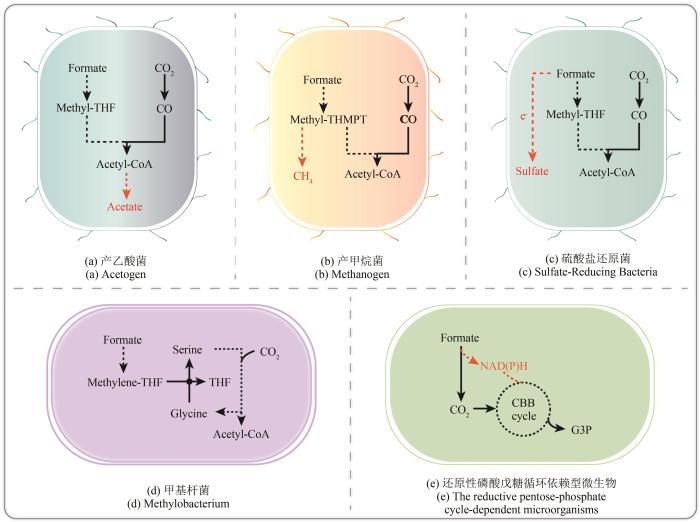
... 产乙酸菌(acetogen)为专性厌氧微生物,可以利用还原性乙酰辅酶A途径进行CO2固定,在完成终端电子接受和能量代谢的同时,将CO2转化为乙酰辅酶A[20][图1(a)].甲酸作为还原性乙酰辅酶A途径中的重要代谢物之一,对于产乙酸菌的生长与代谢具有重要意义,可以被多种产乙酸菌同化利用,如伍氏醋酸杆菌(Acetobacterium woodii)、永达尔梭菌(Clostridium ljungdahlii)和热醋穆尔氏菌(Moorella thermoacetica)等[20,37].近年来,随着甲酸营养型微生物的开发与研究,产乙酸菌可天然利用甲酸的这一特征引起了研究人员的极大重视.其中,以A. woodii作为产乙酸菌模式菌株,通过定量生理学、转录组学、蛋白质组学和代谢模型计算等策略,分析了菌株的甲酸利用水平与甲酸代谢路径,结果表明A. woodii具有良好的单一营养/混合营养型甲酸利用能力与能量利用效率[21-22],从而充分证明了A. woodii作为甲酸天然利用微生物所具有的巨大应用潜能. ...
... [20,37].近年来,随着甲酸营养型微生物的开发与研究,产乙酸菌可天然利用甲酸的这一特征引起了研究人员的极大重视.其中,以A. woodii作为产乙酸菌模式菌株,通过定量生理学、转录组学、蛋白质组学和代谢模型计算等策略,分析了菌株的甲酸利用水平与甲酸代谢路径,结果表明A. woodii具有良好的单一营养/混合营养型甲酸利用能力与能量利用效率[21-22],从而充分证明了A. woodii作为甲酸天然利用微生物所具有的巨大应用潜能. ...
Formate metabolism in the acetogenic bacterium Acetobacterium woodii
1
2021
... 产乙酸菌(acetogen)为专性厌氧微生物,可以利用还原性乙酰辅酶A途径进行CO2固定,在完成终端电子接受和能量代谢的同时,将CO2转化为乙酰辅酶A[20][图1(a)].甲酸作为还原性乙酰辅酶A途径中的重要代谢物之一,对于产乙酸菌的生长与代谢具有重要意义,可以被多种产乙酸菌同化利用,如伍氏醋酸杆菌(Acetobacterium woodii)、永达尔梭菌(Clostridium ljungdahlii)和热醋穆尔氏菌(Moorella thermoacetica)等[20,37].近年来,随着甲酸营养型微生物的开发与研究,产乙酸菌可天然利用甲酸的这一特征引起了研究人员的极大重视.其中,以A. woodii作为产乙酸菌模式菌株,通过定量生理学、转录组学、蛋白质组学和代谢模型计算等策略,分析了菌株的甲酸利用水平与甲酸代谢路径,结果表明A. woodii具有良好的单一营养/混合营养型甲酸利用能力与能量利用效率[21-22],从而充分证明了A. woodii作为甲酸天然利用微生物所具有的巨大应用潜能. ...
A quantitative metabolic analysis reveals Acetobacterium woodii as a flexible and robust host for formate-based bioproduction
2
2021
... 劣势:在厌氧条件下生长,不利于工业应用;代谢研究尚不完善,代谢改造工具少 | [20-22] |
| 产甲烷菌 | 专性厌氧 | 氢营养型海沼甲烷球菌 | 还原性乙酰辅酶A途径 | 优势:能够天然利用甲酸和CO2合成甲烷,在清洁能源产业方面具有应用潜能 ...
... 产乙酸菌(acetogen)为专性厌氧微生物,可以利用还原性乙酰辅酶A途径进行CO2固定,在完成终端电子接受和能量代谢的同时,将CO2转化为乙酰辅酶A[20][图1(a)].甲酸作为还原性乙酰辅酶A途径中的重要代谢物之一,对于产乙酸菌的生长与代谢具有重要意义,可以被多种产乙酸菌同化利用,如伍氏醋酸杆菌(Acetobacterium woodii)、永达尔梭菌(Clostridium ljungdahlii)和热醋穆尔氏菌(Moorella thermoacetica)等[20,37].近年来,随着甲酸营养型微生物的开发与研究,产乙酸菌可天然利用甲酸的这一特征引起了研究人员的极大重视.其中,以A. woodii作为产乙酸菌模式菌株,通过定量生理学、转录组学、蛋白质组学和代谢模型计算等策略,分析了菌株的甲酸利用水平与甲酸代谢路径,结果表明A. woodii具有良好的单一营养/混合营养型甲酸利用能力与能量利用效率[21-22],从而充分证明了A. woodii作为甲酸天然利用微生物所具有的巨大应用潜能. ...
Molecular biology of methanogens
2
1992
... 劣势:在厌氧条件下生长,不利于工业应用;代谢研究尚不完善,代谢改造工具少 | [23-26] |
| 硫酸盐还原菌 | 专型厌氧 | 普通脱硫弧菌 | 还原性乙酰辅酶A途径 | 优势:具有非常高的氢化酶活性,能够天然利用甲酸产氢,在清洁能源产业方面具有应用潜能 ...
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Formate-dependent H2 production by the mesophilic methanogen Methanococcus maripaludis
1
2008
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Genetic systems for hydrogenotrophic methanogens
1
2011
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Metabolic processes of Methanococcus maripaludis and potential applications
2
2016
... 劣势:在厌氧条件下生长,不利于工业应用;代谢研究尚不完善,代谢改造工具少 | [23-26] |
| 硫酸盐还原菌 | 专型厌氧 | 普通脱硫弧菌 | 还原性乙酰辅酶A途径 | 优势:具有非常高的氢化酶活性,能够天然利用甲酸产氢,在清洁能源产业方面具有应用潜能 ...
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Sulfate-reducing bacteria as new microorganisms for biological hydrogen production
3
2013
... 劣势:厌氧条件下生长,不利于工业应用;代谢研究尚不完善,代谢改造工具少 | [27-29] |
| 甲基杆菌 | 好氧或 兼性厌氧 | 扭脱甲基杆菌AM1 | 丝氨酸循环 | 优势:能够天然利用甲酸、CO2与甲醇等一碳底物进行细胞生长;模式菌株的研究与改造相对充分,具有一定的改造潜能 ...
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
... [27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
One-carbon substrate-based biohydrogen production: microbes, mechanism, and productivity
1
2015
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
Desulfovibrio vulgaris growth coupled to formate-driven H2 production
2
2015
... 劣势:厌氧条件下生长,不利于工业应用;代谢研究尚不完善,代谢改造工具少 | [27-29] |
| 甲基杆菌 | 好氧或 兼性厌氧 | 扭脱甲基杆菌AM1 | 丝氨酸循环 | 优势:能够天然利用甲酸、CO2与甲醇等一碳底物进行细胞生长;模式菌株的研究与改造相对充分,具有一定的改造潜能 ...
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
Structure prediction and analysis of MxaF from obligate, facultative and restricted facultative methylobacterium
2
2012
... 劣势:所具有的甲酸代谢路径的能量利用效率偏低,不利于工业应用 | [30-32] |
| CBB循环 依赖型微生物 | 好氧 | 钩虫贪铜菌H16 | 还原性磷酸戊糖循环 | 优势:具有甲酸脱氢酶,能够利用甲酸作为唯一碳源和能源;模式菌株的研究与改造相对充分,具有一定的应用潜能 ...
... 甲基杆菌属(Methylobacterium)细菌为好氧或兼性厌氧的甲基营养型微生物,能够在甲酸、甲醛、甲醇或甲胺等一碳化合物上进行生长,也能利用二碳、三碳和四碳等多碳化合物进行生长[30][图1(d)].扭脱甲基杆菌AM1(Methylobacterium extorquens AM1)为甲基杆菌的模式菌株,能够利用甲醇作唯一碳源和能源.以往关于M. extorquens AM1的研究,多集中于以甲醇为底物生产聚羟基丁酸酯、有机酸和精细化学品等[49].近年来,随着甲酸为底物的优势逐步凸显出来,M. extorquens AM1可通过丝氨酸循环利用甲酸的特性也引起了研究人员的重视[31].特别是对M. extorquens AM1的5,10-次甲基四氢叶酸环化酶的研究,为甲酸同化分子机制的研究奠定了生物化学基础[50].另外,通过甲醇与甲酸的耦合利用,提高了M. extorquens AM1的甲羟戊酸产量、产率和甲醇消耗速率[32],充分体现了M. extorquens AM1代谢利用甲酸的优势,为一碳化合物转化为高附加值化学品提供了一种切实可行的途径. ...
Formate as the main branch point for methylotrophic metabolism in Methylobacterium extorquens AM1
2
2008
... 甲基杆菌属(Methylobacterium)细菌为好氧或兼性厌氧的甲基营养型微生物,能够在甲酸、甲醛、甲醇或甲胺等一碳化合物上进行生长,也能利用二碳、三碳和四碳等多碳化合物进行生长[30][图1(d)].扭脱甲基杆菌AM1(Methylobacterium extorquens AM1)为甲基杆菌的模式菌株,能够利用甲醇作唯一碳源和能源.以往关于M. extorquens AM1的研究,多集中于以甲醇为底物生产聚羟基丁酸酯、有机酸和精细化学品等[49].近年来,随着甲酸为底物的优势逐步凸显出来,M. extorquens AM1可通过丝氨酸循环利用甲酸的特性也引起了研究人员的重视[31].特别是对M. extorquens AM1的5,10-次甲基四氢叶酸环化酶的研究,为甲酸同化分子机制的研究奠定了生物化学基础[50].另外,通过甲醇与甲酸的耦合利用,提高了M. extorquens AM1的甲羟戊酸产量、产率和甲醇消耗速率[32],充分体现了M. extorquens AM1代谢利用甲酸的优势,为一碳化合物转化为高附加值化学品提供了一种切实可行的途径. ...
... 丝氨酸循环(serine cycle)是多种甲基营养型微生物(如M. extorquens AM1[31])同化利用甲酸的代谢途径.该循环包含了二碳、三碳及四碳化合物之间的相互转化[图3(b)]:首先是四氢叶酸(tetrahydrofolic acid,简称THF)循环,即甲酸分子在甲酸四氢叶酸连接酶、次甲基四氢叶酸环水解酶和亚甲基四氢叶酸脱氢酶的先后作用下,生成5,10-亚甲基四氢叶酸,随后5,10-亚甲基四氢叶酸的甲基转移到二碳化合物甘氨酸上,生成三碳化合物丝氨酸;其次,丝氨酸依次转化为羟基丙酮酸、甘油酸、2-磷酸甘油酸与磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP),随后PEP经羧化作用固定1分子CO2生成四碳化合物草酰乙酸;然后,草酰乙酸依次转化为苹果酸与苹果酰辅酶A;最后,苹果酰辅酶A裂解为两个二碳化合物乙酰辅酶A与乙醛酸,其中乙醛酸能够再次转化为甘氨酸用于维持丝氨酸循环.综上所述,每轮丝氨酸循环能够同化1分子甲酸并固定1分子CO2,最终生成1分子乙酰辅酶A,共计消耗3分子NAD(P)H与3分子ATP.与甲酸氧化供能相比,丝氨酸循环可以同化甲酸并固定CO2,不仅避免了碳损失,而且还能在一定程度上减少能量浪费.通过在E. coli中引入异源的天然丝氨酸循环,同时结合实验室适应性进化策略,获得了能够同化利用甲酸合成乙醇的工程菌株[73],从而验证了丝氨酸循环在模式微生物中同化利用甲酸的可行性.然而,天然丝氨酸循环所涉及的酶和代谢反应比较多,且ATP利用效率仍然比较低,因此,利用该循环同化甲酸的微生物,目标代谢物的产量偏低,目前还不适合用于工业化生产. ...
Sodium formate redirects carbon flux and enhances heterologous mevalonate production in Methylobacterium extorquens AM1
2
2023
... 劣势:所具有的甲酸代谢路径的能量利用效率偏低,不利于工业应用 | [30-32] |
| CBB循环 依赖型微生物 | 好氧 | 钩虫贪铜菌H16 | 还原性磷酸戊糖循环 | 优势:具有甲酸脱氢酶,能够利用甲酸作为唯一碳源和能源;模式菌株的研究与改造相对充分,具有一定的应用潜能 ...
... 甲基杆菌属(Methylobacterium)细菌为好氧或兼性厌氧的甲基营养型微生物,能够在甲酸、甲醛、甲醇或甲胺等一碳化合物上进行生长,也能利用二碳、三碳和四碳等多碳化合物进行生长[30][图1(d)].扭脱甲基杆菌AM1(Methylobacterium extorquens AM1)为甲基杆菌的模式菌株,能够利用甲醇作唯一碳源和能源.以往关于M. extorquens AM1的研究,多集中于以甲醇为底物生产聚羟基丁酸酯、有机酸和精细化学品等[49].近年来,随着甲酸为底物的优势逐步凸显出来,M. extorquens AM1可通过丝氨酸循环利用甲酸的特性也引起了研究人员的重视[31].特别是对M. extorquens AM1的5,10-次甲基四氢叶酸环化酶的研究,为甲酸同化分子机制的研究奠定了生物化学基础[50].另外,通过甲醇与甲酸的耦合利用,提高了M. extorquens AM1的甲羟戊酸产量、产率和甲醇消耗速率[32],充分体现了M. extorquens AM1代谢利用甲酸的优势,为一碳化合物转化为高附加值化学品提供了一种切实可行的途径. ...
Genome sequence of the bioplastic-producing "Knallgas" bacterium Ralstonia eutropha H16
2
2006
... 劣势:甲酸的氧化供能存在能量浪费,因此微生物的细胞生长速率与工业生产潜能均偏低 | [33-36] |
注:CBB循环为还原性磷酸戊糖循环的别称. ...
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
Genomic view of energy metabolism in Ralstonia eutropha H16
1
2008
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
Synthetic biology toolkit for engineering Cupriviadus necator H16 as a platform for CO2 valorization
1
2021
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
Improving growth of Cupriavidus necator H16 on formate using adaptive laboratory evolution-informed engineering
3
2023
... 劣势:甲酸的氧化供能存在能量浪费,因此微生物的细胞生长速率与工业生产潜能均偏低
[33-36] | 注:CBB循环为还原性磷酸戊糖循环的别称. ...
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Autotrophy at the thermodynamic limit of life: a model for energy conservation in acetogenic bacteria
1
2014
... 产乙酸菌(acetogen)为专性厌氧微生物,可以利用还原性乙酰辅酶A途径进行CO2固定,在完成终端电子接受和能量代谢的同时,将CO2转化为乙酰辅酶A[20][图1(a)].甲酸作为还原性乙酰辅酶A途径中的重要代谢物之一,对于产乙酸菌的生长与代谢具有重要意义,可以被多种产乙酸菌同化利用,如伍氏醋酸杆菌(Acetobacterium woodii)、永达尔梭菌(Clostridium ljungdahlii)和热醋穆尔氏菌(Moorella thermoacetica)等[20,37].近年来,随着甲酸营养型微生物的开发与研究,产乙酸菌可天然利用甲酸的这一特征引起了研究人员的极大重视.其中,以A. woodii作为产乙酸菌模式菌株,通过定量生理学、转录组学、蛋白质组学和代谢模型计算等策略,分析了菌株的甲酸利用水平与甲酸代谢路径,结果表明A. woodii具有良好的单一营养/混合营养型甲酸利用能力与能量利用效率[21-22],从而充分证明了A. woodii作为甲酸天然利用微生物所具有的巨大应用潜能. ...
Metabolic, phylogenetic, and ecological diversity of the methanogenic Archaea
2
2008
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
... [38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
氢营养型产甲烷代谢途径研究进展
1
2020
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Recent advances in hydrogenotrophic methanogenesis
1
2020
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Methanogenic Archaea: ecologically relevant differences in energy conservation
1
2008
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Protein complexing in a methanogen suggests electron bifurcation and electron delivery from formate to heterodisulfide reductase
1
2010
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
CRISPR-Cas9 toolkit for genome editing in an autotrophic CO2-fixing methanogenic archaeon
1
2022
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
Efficient CRISPR/Cas12a-based genome-editing toolbox for metabolic engineering in Methanococcus maripaludis
1
2022
... 产甲烷菌(methanogen)是一种专性厌氧的古菌,可将CO2、甲酸、乙酸或含甲基的简单化合物等还原为CH4,并从中获取能量[23][图1(b)].根据代谢底物类型的不同,可将产甲烷菌分为氢营养型、甲基营养型和乙酸营养型[38].其中,氢营养型产甲烷菌具有最高的产能效率[39],部分氢营养型产甲烷菌可以利用甲酸作为主要的电子供体,将CO2还原为CH4进行细胞生长[38,40-41].氢营养型海沼甲烷球菌(Methanococcus maripaludis)作为产甲烷菌的模式菌株,不仅能利用氢气与甲酸作为电子供体,还能以高转化率将甲酸转化为氢气[24].目前,基于M. maripaludis已经开发出了一系列分子遗传学操作工具与技术[25-26],如基于CRISPR/Cas9系统[42]与CRISPR/Cas12a系统[43]的基因编辑工具,充分显示出了产甲烷菌进行代谢工程改造的潜能与应用优势. ...
The genome sequence of the anaerobic, sulfate-reducing bacterium Desulfovibrio vulgaris hildenborough
1
2004
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
Carbon assimilation pathways in sulfate reducing bacteria. Formate, carbon dioxide, carbon monoxide, and acetate assimilation by Desulfovibrio baarsii
1
1984
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
The reductive glycine pathway allows autotrophic growth of Desulfovibrio desulfuricans
2
2020
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
... 还原性甘氨酸途径(reductive glycine pathway,简称为rGly途径),主要存在于硫酸盐还原菌(如D. desulfuricans)[46]和产乙酸菌(如梭状芽孢杆菌Clostridium drakei)[77]中.该途径主要包括三个模块[图3(d)]:首先,由甲酸到5,10-亚甲基四氢叶酸的转化;其次,5,10-亚甲基四氢叶酸在甘氨酸裂解系统(glycine cleavage system,GCS)的催化下,固定1分子CO2生成甘氨酸;最后,甘氨酸可通过两种路线转化为丙酮酸,一种为还原甘氨酸(reductive glycine,RG)路线,即5,10-亚甲基四氢叶酸的甲基转移到甘氨酸上生成丝氨酸,随后丝氨酸脱氨基形成丙酮酸,另一种为甘氨酸还原酶(glycine reductase,GR)路线,即甘氨酸依次转化为乙酰磷酸、乙酸与乙酰辅酶A,随后固定1分子CO2生成丙酮酸.综上所述,依赖于RG路线的rGly途径能够同化2分子甲酸和1分子CO2,最终生成1分子丙酮酸,共计消耗3分子NAD(P)H与2分子ATP;依赖于GR路线的rGly途径能够同化1分子甲酸和2分子CO2,最终生成1分子丙酮酸,共计消耗2分子NAD(P)H与1分子ATP.rGly途径是一种能够高效同化甲酸的代谢路径,具有许多优势:rGly途径对ATP的利用效率仅次于WL途径;rGly途径为线性途径,降低了代谢改造的难度,且多种微生物具有内源性的rGly途径[78];rGly途径与中心代谢途径的重叠较少,因此异源引入或改造内源rGly途径对本源代谢网络的干扰比较小;与丝氨酸循环和WL途径相比,rGly途径具有更高的代谢物灵活性,可以进一步转化为多种化学品.基于上述优势,rGly途径成为目前最具发展潜力的甲酸代谢路径,与此同时,对路径关键酶GCS的深入研究[79-80],有望进一步优化rGly途径.然而,对于具有天然rGly途径的微生物研究仍然较少,还不能满足现阶段甲酸生物经济的发展要求. ...
Complete genome sequence of Desulfarculus baarsii type strain (2st14T)
1
2010
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
Electron transfer pathways of formate-driven H2 production in Desulfovibrio
1
2016
... 硫酸盐还原菌(sulfate-reducing bacteria,SRB)为专性厌氧微生物,能够以有机物、氢气等作为电子供体,以硫酸盐、亚硫酸盐、硫代硫酸盐等作为末端电子受体,通过异化作用获取能量来进行细胞生长[图1(c)].部分SRB可以利用甲酸作碳源或电子供体,如普通脱硫弧菌(Desulfovibrio vulgaris)[44]、巴氏脱硫弧菌(Desulfovibrio baarsii)[45]、脱硫脱硫弧菌(Desulfovibrio desulfuricans)[46]和巴氏脱硫菌(Desulfarculus baarsii)[47]等.在SRB可利用的众多底物中,甲酸被认为是生物制氢的最佳底物之一[27-28].以D. vulgaris作为SRB的模式菌株,验证了以甲酸为底物驱动产氢的可行性[27,29,48],证明了SRB在生物制氢方面所具有的优势与未来发展空间. ...
Current advance in bioconversion of methanol to chemicals
1
2018
... 甲基杆菌属(Methylobacterium)细菌为好氧或兼性厌氧的甲基营养型微生物,能够在甲酸、甲醛、甲醇或甲胺等一碳化合物上进行生长,也能利用二碳、三碳和四碳等多碳化合物进行生长[30][图1(d)].扭脱甲基杆菌AM1(Methylobacterium extorquens AM1)为甲基杆菌的模式菌株,能够利用甲醇作唯一碳源和能源.以往关于M. extorquens AM1的研究,多集中于以甲醇为底物生产聚羟基丁酸酯、有机酸和精细化学品等[49].近年来,随着甲酸为底物的优势逐步凸显出来,M. extorquens AM1可通过丝氨酸循环利用甲酸的特性也引起了研究人员的重视[31].特别是对M. extorquens AM1的5,10-次甲基四氢叶酸环化酶的研究,为甲酸同化分子机制的研究奠定了生物化学基础[50].另外,通过甲醇与甲酸的耦合利用,提高了M. extorquens AM1的甲羟戊酸产量、产率和甲醇消耗速率[32],充分体现了M. extorquens AM1代谢利用甲酸的优势,为一碳化合物转化为高附加值化学品提供了一种切实可行的途径. ...
Structural insight into a molecular mechanism of methenyltetrahydrofolate cyclohydrolase from Methylobacterium extorquens AM1
1
2022
... 甲基杆菌属(Methylobacterium)细菌为好氧或兼性厌氧的甲基营养型微生物,能够在甲酸、甲醛、甲醇或甲胺等一碳化合物上进行生长,也能利用二碳、三碳和四碳等多碳化合物进行生长[30][图1(d)].扭脱甲基杆菌AM1(Methylobacterium extorquens AM1)为甲基杆菌的模式菌株,能够利用甲醇作唯一碳源和能源.以往关于M. extorquens AM1的研究,多集中于以甲醇为底物生产聚羟基丁酸酯、有机酸和精细化学品等[49].近年来,随着甲酸为底物的优势逐步凸显出来,M. extorquens AM1可通过丝氨酸循环利用甲酸的特性也引起了研究人员的重视[31].特别是对M. extorquens AM1的5,10-次甲基四氢叶酸环化酶的研究,为甲酸同化分子机制的研究奠定了生物化学基础[50].另外,通过甲醇与甲酸的耦合利用,提高了M. extorquens AM1的甲羟戊酸产量、产率和甲醇消耗速率[32],充分体现了M. extorquens AM1代谢利用甲酸的优势,为一碳化合物转化为高附加值化学品提供了一种切实可行的途径. ...
From formic acid to single-cell protein: genome-scale revealing the metabolic network of Paracoccus communis MA5
1
2022
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
Formate-dependent autotrophic growth in Sinorhizobium meliloti
1
2008
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
Nitrification expanded: discovery, physiology and genomics of a nitrite-oxidizing bacterium from the Phylum chloroflexi
1
2012
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
RubisCO-based CO2 fixation and C1 metabolism in the actinobacterium Pseudonocardia dioxanivorans?CB1190
1
2013
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
Genomic insights into metabolic versatility of a lithotrophic sulfur-oxidizing diazotrophic Alphaproteobacterium Azospirillum thiophilum
1
2016
... 除了上述天然甲酸利用微生物之外,近年来研究人员逐步分离与鉴定出更多新的天然甲酸利用微生物,如能够通过丝氨酸循环同化甲酸合成单细胞蛋白的共生副球菌MA5(Paracoccus communis MA5)[51],这在一定程度上拓宽了甲酸利用微生物的应用范围.另外,除了还原性乙酰辅酶A途径、丝氨酸循环以及还原性甘氨酸途径之外,部分天然甲酸利用微生物还可以通过还原性磷酸戊糖循环利用甲酸,如C. necator H16[33-34]、苜蓿中华根瘤菌(Sinorhizobium meliloti)[52]、亚硝酸盐氧化细菌(Nitrolancetus hollandicus)[53]、嗜二氧杂环乙烷假诺卡氏菌CB1190(Pseudonocardia dioxanivorans CB1190)[54]和嗜硫偶氮螺旋菌(Azospirillum thiophilum)[55]等,它们都具有甲酸脱氢酶,能够在甲酸氧化为CO2的同时产生还原力,并通过还原性磷酸戊糖循环固定CO2进行细胞生长与代谢[图1(e)].C. necator H16作为这一类型微生物的模式菌株,已经开发出一系列相应的分子遗传学操作工具与技术,并结合代谢工程改造策略将其应用于开发微生物细胞工厂生产多种代谢产物[35-36],极大地拓宽了生物固碳与甲酸生物经济的发展空间. ...
Synthetic formatotrophs for one-carbon biorefinery
3
2021
... 通过实验室适应性进化与培养条件优化等策略提高菌株对甲酸的耐受性
[17-18,56-67] | | 酿酒酵母 | rTHF-rgcv途径 的关键模块 | 遗传背景清晰; ...
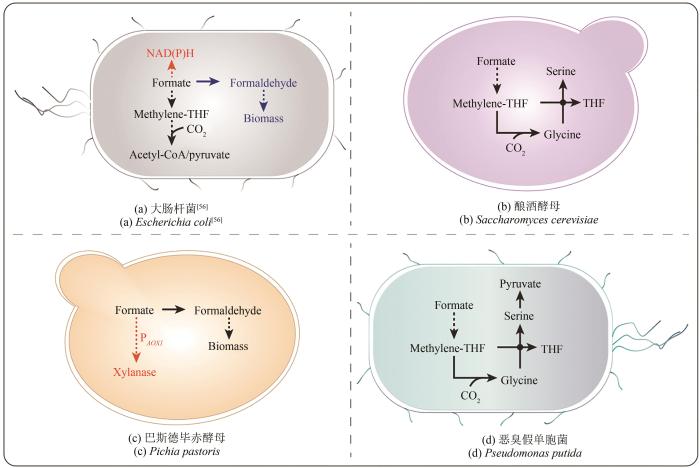
... E. coli作为常用的原核模式微生物,近年来已有多项研究聚焦于改造E. coli利用甲酸.在这些研究中,通过构建甲酸利用路径[图2(a)],并结合代谢工程策略与实验室适应性进化等策略,使得E. coli能够利用甲酸(或甲酸与CO2)进行细胞生长[56],充分体现了实验室改造模式微生物利用甲酸的可行性.然而,这种甲酸营养型E. coli的生长速率与细胞密度显著低于使用常规碳源培养的E. coli,主要原因在于甲酸营养型E. coli对甲酸的同化效率与耐受性均偏低. ...
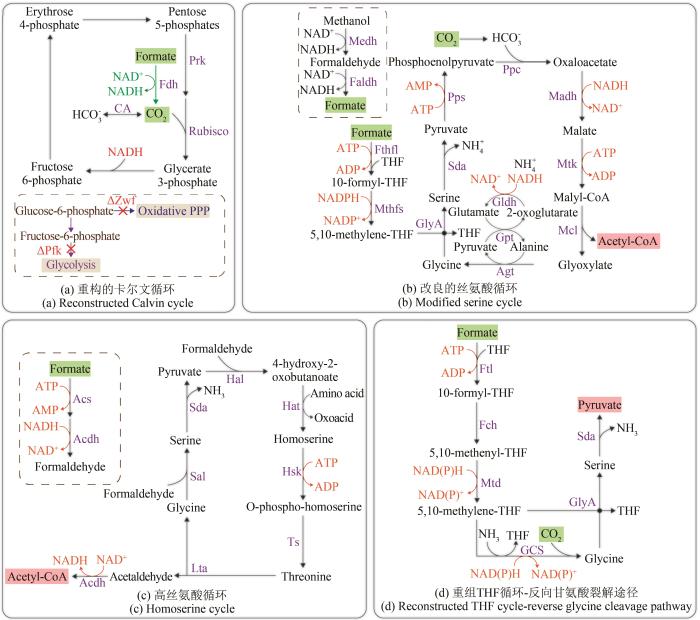
... 基于丝氨酸循环,在E. coli中构建了高丝氨酸循环(homoserine cycle)[59][图4(c)].首先,甲酸在乙酰辅酶A合成酶和乙醛脱氢酶的作用下转化为甲醛,甲醛作为一种高活性化合物,更易于被同化进入代谢系统[56,85];其次,在丝氨酸醛缩酶的作用下,甲醛与甘氨酸反应生成丝氨酸,随之丝氨酸脱氨形成丙酮酸;然后,另一分子甲醛与丙酮酸在4-羟基-2-氧代丁酸(4-hydroxy-2-oxobutanoate,HOB)醛缩酶的作用下,生成非天然代谢物HOB;随后,氨基被结合到HOB上生成高丝氨酸,高丝氨酸在高丝氨酸激酶与苏氨酸合成酶的作用下转化为苏氨酸;最后,在苏氨酸醛缩酶的催化下,苏氨酸裂解为甘氨酸与乙醛.其中,甘氨酸重新进入循环,而乙醛分子经乙醛脱氢酶的催化,生成乙酰辅酶A并输出循环.综上所述,每轮高丝氨酸循环能够同化2分子甲酸,最终生成1分子乙酰辅酶A,共计消耗1分子NADH与3分子ATP.高丝氨酸循环是在改良丝氨酸循环的基础上,对天然丝氨酸循环进行的更深层的优化,具有明显的优势:以甲醛替代CO2进入同化路径,改善了羧化反应所造成的限速作用,减少了还原力的使用,有利于支持更高的生物量产量;该循环路径所涉及的酶均为E. coli的内源酶,保证了酶的催化活性;相比于丝氨酸循环,该循环所包含的酶促反应较少,且与中心代谢途径的重叠也较少[59].然而,甲醛与高丝氨酸对E. coli都具有一定的毒性作用.因此,深入探究E. coli的毒性机理与解毒机制[86-88],对于进一步优化高丝氨酸循环具有重要意义. ...
Conversion of Escherichia coli to generate all biomass carbon from CO2
2
2019
... 通过在E. coli中重构卡尔文循环,首次实现了异养微生物向自养微生物的转化[57].通过敲除糖酵解途径中的磷酸果糖激酶和戊糖磷酸途径中的葡萄糖-6-磷酸脱氢酶,并异源表达核酮糖-1,5-二磷酸羧化酶、磷酸核酮糖激酶、碳酸酐酶和甲酸脱氢酶,实现了在E. coli中重构卡尔文循环[图4(a)].重构的卡尔文循环(reconstructed Calvin cycle)可以固定CO2合成生物量,同时氧化甲酸实现供能.在此基础上,将该循环与实验室适应性进化相结合,进化获得了自养型E. coli,这种基于甲酸供能的生物固碳模式在甲酸生物经济发展和未来工业生物制造方面具有极大的潜力[81].此外,随着近年来电化学法还原CO2生产甲酸[82-84]的深入研究,该自养系统中由于甲酸氧化速率高于固碳速率而导致的CO2净排放也有望得到优化与解决. ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
A modified serine cycle in Escherichia coli coverts methanol and CO2 to two-carbon compounds
1
2018
... 基于源自M. extorquens AM1的天然丝氨酸循环,通过在E. coli中进行表达与改良丝氨酸循环,获得的工程菌株能够利用甲醇、甲酸和CO2合成乙酰辅酶A[58].改良的丝氨酸循环(modifed serine cycle)同化甲酸的过程主要分为以下几个步骤[图4(b)]:首先,甲酸在甲酸四氢叶酸连接酶和5,10-亚甲基四氢叶酸合成酶的作用下转化为5,10-亚甲基四氢叶酸,随后在丝氨酸羟甲基转移酶的催化下,5,10-亚甲基四氢叶酸的甲基转移至甘氨酸上,生成丝氨酸;其次,丝氨酸在丝氨酸脱氨酶的催化下脱氨生成丙酮酸,再经PEP合成酶、PEP羧化酶的作用,固定1分子CO2生成草酰乙酸;再次,草酰乙酸在苹果酸脱氢酶和苹果酸硫激酶的作用下转化为苹果酰辅酶A;最后,苹果酰辅酶A经苹果酰辅酶A裂解酶的催化,裂解为乙醛酸与乙酰辅酶A.其中,乙酰辅酶A作为该循环的输出产物,而乙醛酸则转化为甘氨酸用于完成新一轮循环.综上所述,当甲酸作为底物时,改良的丝氨酸循环与天然丝氨酸循环一致,每轮循环能够同化1分子甲酸并固定1分子CO2,最终生成1分子乙酰辅酶A,共计消耗3分子NAD(P)H与3分子ATP.相比于天然丝氨酸循环,改良的丝氨酸循环具有一定生物优势.通过甲醇脱氢酶和甲醛脱氢酶,不仅简化了甲醇转化为甲酸的路径,而且还利用丙氨酸-乙醛酸转氨酶、谷氨酸-丙酮酸转氨酶和谷氨酸脱氢酶进行氨同化,以丙氨酸代替丝氨酸成为氨基供体,从而避免了有毒中间代谢物羟基丙酮酸的生成.然而,改良的丝氨酸循环路径比较复杂,涉及多个酶促反应,且与中心代谢途径的重叠较多.因此,仍存在ATP利用效率偏低的问题,不利于进一步的代谢工程改造与工业应用. ...
An optimized methanol assimilation pathway relying on promiscuous formaldehyde-condensing aldolases in E. coli
2
2020
... 基于丝氨酸循环,在E. coli中构建了高丝氨酸循环(homoserine cycle)[59][图4(c)].首先,甲酸在乙酰辅酶A合成酶和乙醛脱氢酶的作用下转化为甲醛,甲醛作为一种高活性化合物,更易于被同化进入代谢系统[56,85];其次,在丝氨酸醛缩酶的作用下,甲醛与甘氨酸反应生成丝氨酸,随之丝氨酸脱氨形成丙酮酸;然后,另一分子甲醛与丙酮酸在4-羟基-2-氧代丁酸(4-hydroxy-2-oxobutanoate,HOB)醛缩酶的作用下,生成非天然代谢物HOB;随后,氨基被结合到HOB上生成高丝氨酸,高丝氨酸在高丝氨酸激酶与苏氨酸合成酶的作用下转化为苏氨酸;最后,在苏氨酸醛缩酶的催化下,苏氨酸裂解为甘氨酸与乙醛.其中,甘氨酸重新进入循环,而乙醛分子经乙醛脱氢酶的催化,生成乙酰辅酶A并输出循环.综上所述,每轮高丝氨酸循环能够同化2分子甲酸,最终生成1分子乙酰辅酶A,共计消耗1分子NADH与3分子ATP.高丝氨酸循环是在改良丝氨酸循环的基础上,对天然丝氨酸循环进行的更深层的优化,具有明显的优势:以甲醛替代CO2进入同化路径,改善了羧化反应所造成的限速作用,减少了还原力的使用,有利于支持更高的生物量产量;该循环路径所涉及的酶均为E. coli的内源酶,保证了酶的催化活性;相比于丝氨酸循环,该循环所包含的酶促反应较少,且与中心代谢途径的重叠也较少[59].然而,甲醛与高丝氨酸对E. coli都具有一定的毒性作用.因此,深入探究E. coli的毒性机理与解毒机制[86-88],对于进一步优化高丝氨酸循环具有重要意义. ...
... [59].然而,甲醛与高丝氨酸对E. coli都具有一定的毒性作用.因此,深入探究E. coli的毒性机理与解毒机制[86-88],对于进一步优化高丝氨酸循环具有重要意义. ...
Engineered assimilation of exogenous and endogenous formate in Escherichia coli
1
2017
... 路径模块化优化作为一种理性的代谢工程策略,通过将甲酸同化路径划分为几个代谢模块,并利用相应的营养缺陷型菌株测试各个模块的活性与限制性瓶颈,通过解除瓶颈最终实现甲酸同化路径的整体优化与适配[96][图6(e)].例如,借助模块化工程策略,通过组合rTHF模块与丝氨酸-苏氨酸循环,验证了甲酸同化在E. coli中的代谢可行性[60].首先,通过在一碳与甘氨酸营养缺陷型菌株中过表达异源Ftl,甲酸能够回补菌株的一碳营养缺陷,从而证明了一碳模块的活性;其次,通过在丝氨酸营养缺陷型菌株中过表达rTHF模块的Fch/ Mtd双功能酶(FolD)与GlyA,工程菌株实现了在甲酸与甘氨酸上进行生长;最后,通过在丝氨酸营养缺陷型菌株中引入丝氨酸-苏氨酸循环,使工程菌株能够在甲酸与葡萄糖上进行细胞生长,不再需要提供外源甘氨酸.上述研究为后续甲酸同化路径的构建提供了重要参考,模块化工程不仅实现了甲酸营养型E. coli的构建[17],而且还完成了P. putida甲酸代谢改造[70]等,从而拓展了同化甲酸的底盘微生物. ...
Electrical-biological hybrid system for CO2 reduction
2
2018
| [61] |
| Escherichia coli | Ftl、Fch、Mtd(源自Methylobacterium extorquens AM1) ...
... 甲酸同化路径的构建方式主要包括两种:一种是将异源路径基因与微生物的内源基因进行理性组合;另一种是仅对微生物内源基因进行系统优化.然而,上述两种构建方式均会影响微生物的本源代谢网络,从而影响甲酸同化效率.因此,需要对路径基因的表达水平进行合理优化[图6(a)].采用的策略主要包括两种:①选择合适的异源基因,用于构建甲酸同化路径.例如,当在E. coli中引入甲酸四氢叶酸连接酶(Ftl)、次甲基四氢叶酸环水解酶(Fch)和亚甲基四氢叶酸脱氢酶(Mtd)等基因构建THF循环[图4(d)]时,同时将源自C. ljungdahlii和A. woodie的基因操纵子Cl和Aw分别引入丝氨酸营养缺陷型菌株中,结果显示Cl操纵子可以回补菌株的丝氨酸营养缺陷,使其以甲酸为碳源进行生长,但是Aw操纵子却不能实现上述效果.上述结果表明,Cl操纵子有利于异源Ftl、Fch和Mtd以及内源丝氨酸羟甲基转移酶(GlyA)的催化作用,实现甲酸到丝氨酸的转化[61].然而,上述研究所构建的THF循环对甲酸的同化效率明显低于源自M. extorquens的THF循环[62-63],这也进一步证明了选择合适的异源基因对于构建甲酸同化路径的重要性.②优化路径基因水平,实现路径与宿主的最优适配.例如,采用高拷贝数、中拷贝数和低拷贝数的质粒,在E. coli中表达Ftl、Fch、Mtd与两种甲酸脱氢酶,结果显示含有低拷贝数质粒的菌株生长速率明显高于中含有高拷贝数的菌株.上述结果表明,低拷贝数质粒有利于实现菌株路径基因的最优表达[18].除了利用不同拷贝数的质粒外,启动子工程也可以用于优化路径基因表达水平.例如,通过将不同启动子组合的质粒pC1(表达Ftl、Fch、Mtd)和pC2(表达GCS)分别导入E. coli甘氨酸营养缺陷型菌株,结果显示当采用弱启动子时菌株生长最好.上述结果表明,弱启动子组合有利于实现路径基因的最优表达[16].另外,值得注意的是,虽然使用质粒过表达路径基因相对简捷,但是过多质粒的使用会影响微生物的生长,相比而言基因组的过表达更加稳定.例如,首先在E. coli的基因组上过表达GCS,随后导入rTHF循环,获得了菌株E. coli RG3.与使用质粒过表达GCS的菌株E. coli RG2相比,E. coli RG3具有更高的生长速率与产物产率[63]. ...
In vivo assimilation of one-carbon via a synthetic reductive glycine pathway in Escherichia coli
2
2018
| [62] |
| Escherichia coli | Ftl、Fch、Mtd(源自Methylobacterium extorquens CM4) ...
... 甲酸同化路径的构建方式主要包括两种:一种是将异源路径基因与微生物的内源基因进行理性组合;另一种是仅对微生物内源基因进行系统优化.然而,上述两种构建方式均会影响微生物的本源代谢网络,从而影响甲酸同化效率.因此,需要对路径基因的表达水平进行合理优化[图6(a)].采用的策略主要包括两种:①选择合适的异源基因,用于构建甲酸同化路径.例如,当在E. coli中引入甲酸四氢叶酸连接酶(Ftl)、次甲基四氢叶酸环水解酶(Fch)和亚甲基四氢叶酸脱氢酶(Mtd)等基因构建THF循环[图4(d)]时,同时将源自C. ljungdahlii和A. woodie的基因操纵子Cl和Aw分别引入丝氨酸营养缺陷型菌株中,结果显示Cl操纵子可以回补菌株的丝氨酸营养缺陷,使其以甲酸为碳源进行生长,但是Aw操纵子却不能实现上述效果.上述结果表明,Cl操纵子有利于异源Ftl、Fch和Mtd以及内源丝氨酸羟甲基转移酶(GlyA)的催化作用,实现甲酸到丝氨酸的转化[61].然而,上述研究所构建的THF循环对甲酸的同化效率明显低于源自M. extorquens的THF循环[62-63],这也进一步证明了选择合适的异源基因对于构建甲酸同化路径的重要性.②优化路径基因水平,实现路径与宿主的最优适配.例如,采用高拷贝数、中拷贝数和低拷贝数的质粒,在E. coli中表达Ftl、Fch、Mtd与两种甲酸脱氢酶,结果显示含有低拷贝数质粒的菌株生长速率明显高于中含有高拷贝数的菌株.上述结果表明,低拷贝数质粒有利于实现菌株路径基因的最优表达[18].除了利用不同拷贝数的质粒外,启动子工程也可以用于优化路径基因表达水平.例如,通过将不同启动子组合的质粒pC1(表达Ftl、Fch、Mtd)和pC2(表达GCS)分别导入E. coli甘氨酸营养缺陷型菌株,结果显示当采用弱启动子时菌株生长最好.上述结果表明,弱启动子组合有利于实现路径基因的最优表达[16].另外,值得注意的是,虽然使用质粒过表达路径基因相对简捷,但是过多质粒的使用会影响微生物的生长,相比而言基因组的过表达更加稳定.例如,首先在E. coli的基因组上过表达GCS,随后导入rTHF循环,获得了菌株E. coli RG3.与使用质粒过表达GCS的菌株E. coli RG2相比,E. coli RG3具有更高的生长速率与产物产率[63]. ...
Assimilation of formic acid and CO2 by engineered Escherichia coli equipped with reconstructed one-carbon assimilation pathways
6
2018
... Fdh(源自Candida boidinii) | [63] |
| Escherichia coli | Ftl(源自Clostridium kluyveri) ...
... (Bang等[63]研究的进一步拓展) ...
... 甲酸同化路径的构建方式主要包括两种:一种是将异源路径基因与微生物的内源基因进行理性组合;另一种是仅对微生物内源基因进行系统优化.然而,上述两种构建方式均会影响微生物的本源代谢网络,从而影响甲酸同化效率.因此,需要对路径基因的表达水平进行合理优化[图6(a)].采用的策略主要包括两种:①选择合适的异源基因,用于构建甲酸同化路径.例如,当在E. coli中引入甲酸四氢叶酸连接酶(Ftl)、次甲基四氢叶酸环水解酶(Fch)和亚甲基四氢叶酸脱氢酶(Mtd)等基因构建THF循环[图4(d)]时,同时将源自C. ljungdahlii和A. woodie的基因操纵子Cl和Aw分别引入丝氨酸营养缺陷型菌株中,结果显示Cl操纵子可以回补菌株的丝氨酸营养缺陷,使其以甲酸为碳源进行生长,但是Aw操纵子却不能实现上述效果.上述结果表明,Cl操纵子有利于异源Ftl、Fch和Mtd以及内源丝氨酸羟甲基转移酶(GlyA)的催化作用,实现甲酸到丝氨酸的转化[61].然而,上述研究所构建的THF循环对甲酸的同化效率明显低于源自M. extorquens的THF循环[62-63],这也进一步证明了选择合适的异源基因对于构建甲酸同化路径的重要性.②优化路径基因水平,实现路径与宿主的最优适配.例如,采用高拷贝数、中拷贝数和低拷贝数的质粒,在E. coli中表达Ftl、Fch、Mtd与两种甲酸脱氢酶,结果显示含有低拷贝数质粒的菌株生长速率明显高于中含有高拷贝数的菌株.上述结果表明,低拷贝数质粒有利于实现菌株路径基因的最优表达[18].除了利用不同拷贝数的质粒外,启动子工程也可以用于优化路径基因表达水平.例如,通过将不同启动子组合的质粒pC1(表达Ftl、Fch、Mtd)和pC2(表达GCS)分别导入E. coli甘氨酸营养缺陷型菌株,结果显示当采用弱启动子时菌株生长最好.上述结果表明,弱启动子组合有利于实现路径基因的最优表达[16].另外,值得注意的是,虽然使用质粒过表达路径基因相对简捷,但是过多质粒的使用会影响微生物的生长,相比而言基因组的过表达更加稳定.例如,首先在E. coli的基因组上过表达GCS,随后导入rTHF循环,获得了菌株E. coli RG3.与使用质粒过表达GCS的菌株E. coli RG2相比,E. coli RG3具有更高的生长速率与产物产率[63]. ...
... [63]. ...
... 甲酸同化路径的代谢流通量受到路径关键酶活性的直接影响.因此,结合路径关键酶的催化特点并进行理性改造,能够减少甚至消除路径表达的限制性因素,从而提高甲酸同化路径的催化效率.以rTHF-rgcv途径的关键酶GCS为例,GCS是由四种蛋白质(T/H/P/L)组成的多酶复合体,催化的甘氨酸代谢为可逆反应[图6(b)],但是在E. coli等多数微生物中的GCS更倾向于催化甘氨酸的裂解而非合成,因此,在构建rTHF-rgcv途径的过程中,不仅需要改造GCS的催化反应方向,而且还需要提高GCS的催化活性.例如,通过在E. coli中过表达内源GCS和敲除gcvR基因(抑制编码蛋白T/H/P的基因转录),工程菌株E. coli RG1能够更好地转化甲酸与CO2生成甘氨酸,从而证明了改造GCS有利于提高甲酸同化路径的代谢通量[63]. ...
... 由于甲酸同化路径与微生物的本源代谢网络存在一定的交叉性,所以不可避免地存在着路径代谢流的泄露,因此,需要对甲酸同化路径的竞争路径进行合理阻断.以rTHF-rgcv途径为例[图6(c)],一方面阻断本源代谢网络中能够合成甲酸同化途径中间产物的路径.例如,通过在serA基因(编码3-磷酸甘油酸脱氢酶)缺陷型E. coli中构建rTHF-rgcv途径,促进5,10-亚甲基四氢叶酸的合成,使得rTHF-rgcv途径的丙酮酸合成通量达到了总丙酮酸合成通量的12.9%,而在未敲除serA基因的菌株中仅为7.3%[63].另一方面阻断能够代谢甲酸同化途径中间产物的路径.例如,C. necator能够通过乙醛酸路径同化甘氨酸,即甘氨酸先氧化生成乙醛酸,随后乙醛酸通过甘油酸途径转化为生物量.然而,相比于rTHF-rgcv途径中的丝氨酸路径,乙醛酸路径同化甘氨酸的效率更低,故丝氨酸路径取代乙醛酸路径更有利于菌株在甲酸上生长[16]. ...
Implementation of a reductive route of one-carbon assimilation in Escherichia coli through directed evolution
4
2018
... FolD(Fch/ Mtd)、GCS、GlyA(内源酶) | [64] |
| Escherichia coli | Ftl、Fch、Mtd(源自Methylobacterium extorquens AM1) ...
... (Döring等[64]研究的进一步拓展) ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
... [64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Genetic and biocatalytic basis of formate dependent growth of Escherichia coli strains evolved in continuous culture
2
2022
... FolD(Fch/ Mtd)、GCS、GlyA(内源酶) | [65] |
Escherichia coli ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Optimizing E. coli as a formatotrophic platform for bioproduction via the reductive glycine pathway
2
2023
... Fdh(源自Pseudomonas sp. ) | [66] |
| Saccharomyces cerevisiae | MIS1(Ftl/ Fch/ Mtd)、GCS(内源酶) ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Constructing a synthetic pathway for acetyl-coenzyme A from one-carbon through enzyme design
3
2019
... 通过实验室适应性进化与培养条件优化等策略提高菌株对甲酸的耐受性 | [17-18,56-67] |
| 酿酒酵母 | rTHF-rgcv途径 的关键模块 | 遗传背景清晰; ...
... 通过定向进化P. putida的苯甲酰甲酸脱羧酶,获得了乙醇醛合成酶(glycolaldehyde synthase)[67].在此基础上,构建了一条合成乙酰辅酶A途径(synthetic acetyl‑CoA pathway),该途径仅有3步酶促反应[图5(b)]:首先,在乙醇醛合成酶的作用下,两个甲醛分子缩合生成1分子乙醇醛;其次,在乙酰磷酸合成酶的催化下,实现了乙醇醛到乙酰磷酸的转化;最后,乙酰磷酸在磷酸转乙酰酶的催化下生成乙酰辅酶A.综上所述,该路径能够同化2分子甲醛生成1分子乙酰辅酶A,且不存在ATP与还原力的消耗.合成乙酰辅酶A途径同样为线性反应路径,仅需3种酶即可完成路径催化.然而,该途径尚未被拓展到直接利用甲酸作为路径的起点,尚需参考甲醛酶途径以实现甲酸到甲醛的转化.另外,由于甲醛毒性以及乙醇醛合成酶和乙酰磷酸合成酶对底物的亲和力比较差,因此在E. coli中引入合成乙酰辅酶A途径后,菌株的生物量偏低[67],从而限制了该路径在现阶段的应用范围. ...
... [67],从而限制了该路径在现阶段的应用范围. ...
Enhancing xylanase expression by Komagataella phaffii by formate as carbon source and inducer
3
2022
... 基于菌株的代谢网络,设计并构建更加高效的甲酸利用路径 | [68-69] |
| 恶臭假单胞菌 | rTHF-rgcv途径 | 能够编码多种天然甲酸脱氢酶; ...
... 甲酸营养型微生物的开发与应用在一碳生物炼制方面展现出了巨大潜能.除了E. coli与S. cerevisiae之外,通过改造其他模式微生物进行甲酸代谢,有利于开发更高效的甲酸营养型微生物细胞工厂.巴斯德毕赤酵母(Pichia pastoris)作为天然的甲基营养型酵母,在一碳生物炼制方面有望成为代谢工程改造的关键底盘微生物.甲醇作为诱导物,可以触发P. pastoris的醇氧化酶(alcohol oxidase 1,AOX1)启动子P AOX1 所调控的异源蛋白表达.然而,由于甲醇具有一定的毒性、易燃性和爆炸性,从而限制了P. pastoris在食品与生物医药等产业的大规模应用.与甲醇相比,甲酸被认为是更安全的P AOX1 的诱导物[68].利用甲酸作为诱导物,可以增强P. pastoris木聚糖酶的表达[图2(c)],体现了该系统在异源蛋白表达与安全性方面的潜力[68-69].此外,通过在恶臭假单胞菌(Pseudomonas putida)中引入异源的还原性甘氨酸途径[图2(d)],并结合模块化工程、代谢工程改造与实验室适应性进化等多项策略,获得了能够同化利用甲酸的工程菌株[70],从而证明了P. putida在甲酸利用方面的潜能. ...
... [68-69].此外,通过在恶臭假单胞菌(Pseudomonas putida)中引入异源的还原性甘氨酸途径[图2(d)],并结合模块化工程、代谢工程改造与实验室适应性进化等多项策略,获得了能够同化利用甲酸的工程菌株[70],从而证明了P. putida在甲酸利用方面的潜能. ...
Enhancing xylanase expression of Komagataella phaffii induced by formate through Mit1 co-expression
2
2022
... 基于菌株的代谢网络,设计并构建更加高效的甲酸利用路径 | [68-69] |
| 恶臭假单胞菌 | rTHF-rgcv途径 | 能够编码多种天然甲酸脱氢酶; ...
... 甲酸营养型微生物的开发与应用在一碳生物炼制方面展现出了巨大潜能.除了E. coli与S. cerevisiae之外,通过改造其他模式微生物进行甲酸代谢,有利于开发更高效的甲酸营养型微生物细胞工厂.巴斯德毕赤酵母(Pichia pastoris)作为天然的甲基营养型酵母,在一碳生物炼制方面有望成为代谢工程改造的关键底盘微生物.甲醇作为诱导物,可以触发P. pastoris的醇氧化酶(alcohol oxidase 1,AOX1)启动子P AOX1 所调控的异源蛋白表达.然而,由于甲醇具有一定的毒性、易燃性和爆炸性,从而限制了P. pastoris在食品与生物医药等产业的大规模应用.与甲醇相比,甲酸被认为是更安全的P AOX1 的诱导物[68].利用甲酸作为诱导物,可以增强P. pastoris木聚糖酶的表达[图2(c)],体现了该系统在异源蛋白表达与安全性方面的潜力[68-69].此外,通过在恶臭假单胞菌(Pseudomonas putida)中引入异源的还原性甘氨酸途径[图2(d)],并结合模块化工程、代谢工程改造与实验室适应性进化等多项策略,获得了能够同化利用甲酸的工程菌株[70],从而证明了P. putida在甲酸利用方面的潜能. ...
Integrated rational and evolutionary engineering of genome-reduced Pseudomonas putida strains promotes synthetic formate assimilation
5
2022
... 具有灵活的代谢机制,能够抵抗氧化应激和多种有毒化合物 | 开发更高效的分子遗传操作工具,进一步完善菌株的甲酸代谢网络 | [70] |
注:rTHF-rgcv途径为重组THF循环-反向甘氨酸裂解途径(reconstructed THF cycle-reverse glycine cleavage pathway). ...
... 甲酸营养型微生物的开发与应用在一碳生物炼制方面展现出了巨大潜能.除了E. coli与S. cerevisiae之外,通过改造其他模式微生物进行甲酸代谢,有利于开发更高效的甲酸营养型微生物细胞工厂.巴斯德毕赤酵母(Pichia pastoris)作为天然的甲基营养型酵母,在一碳生物炼制方面有望成为代谢工程改造的关键底盘微生物.甲醇作为诱导物,可以触发P. pastoris的醇氧化酶(alcohol oxidase 1,AOX1)启动子P AOX1 所调控的异源蛋白表达.然而,由于甲醇具有一定的毒性、易燃性和爆炸性,从而限制了P. pastoris在食品与生物医药等产业的大规模应用.与甲醇相比,甲酸被认为是更安全的P AOX1 的诱导物[68].利用甲酸作为诱导物,可以增强P. pastoris木聚糖酶的表达[图2(c)],体现了该系统在异源蛋白表达与安全性方面的潜力[68-69].此外,通过在恶臭假单胞菌(Pseudomonas putida)中引入异源的还原性甘氨酸途径[图2(d)],并结合模块化工程、代谢工程改造与实验室适应性进化等多项策略,获得了能够同化利用甲酸的工程菌株[70],从而证明了P. putida在甲酸利用方面的潜能. ...
... Fdh(内源酶)
[70] | 注:FolD为Fch/ Mtd双功能酶;MIS1为Ftl/ Fch/ Mtd三功能酶. ...
... 路径模块化优化作为一种理性的代谢工程策略,通过将甲酸同化路径划分为几个代谢模块,并利用相应的营养缺陷型菌株测试各个模块的活性与限制性瓶颈,通过解除瓶颈最终实现甲酸同化路径的整体优化与适配[96][图6(e)].例如,借助模块化工程策略,通过组合rTHF模块与丝氨酸-苏氨酸循环,验证了甲酸同化在E. coli中的代谢可行性[60].首先,通过在一碳与甘氨酸营养缺陷型菌株中过表达异源Ftl,甲酸能够回补菌株的一碳营养缺陷,从而证明了一碳模块的活性;其次,通过在丝氨酸营养缺陷型菌株中过表达rTHF模块的Fch/ Mtd双功能酶(FolD)与GlyA,工程菌株实现了在甲酸与甘氨酸上进行生长;最后,通过在丝氨酸营养缺陷型菌株中引入丝氨酸-苏氨酸循环,使工程菌株能够在甲酸与葡萄糖上进行细胞生长,不再需要提供外源甘氨酸.上述研究为后续甲酸同化路径的构建提供了重要参考,模块化工程不仅实现了甲酸营养型E. coli的构建[17],而且还完成了P. putida甲酸代谢改造[70]等,从而拓展了同化甲酸的底盘微生物. ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Design and analysis of metabolic pathways supporting formatotrophic growth for electricity-dependent cultivation of microbes
2
2013
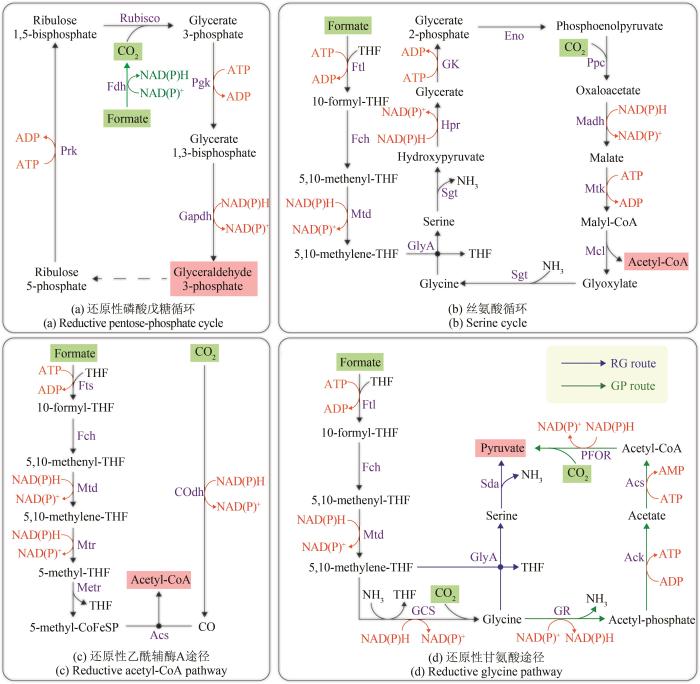
... 还原性磷酸戊糖循环(reductive pentose-phosphate cycle),也称为Calvin-Benson-Bassham(CBB)循环,是唯一已知的能够利用甲酸进行微生物自养生长的天然碳同化途径[71].CBB循环主要包括三个过程[图3(a)]:首先是羧化作用,在关键酶核酮糖-1,5-二磷酸羧化酶/加氧酶的作用下,1分子CO2与1,5-二磷酸核酮糖(ribulose-1,5-bisphosphate,RuBP)分子进行反应,生成一个不稳定的六碳化合物,随之分解为2分子3-磷酸甘油酸;其次是还原作用,2分子3-磷酸甘油酸先转化为2分子1,3-二磷酸甘油酸,随之生成2分子3-磷酸甘油醛;最后是RuBP的再生,在经过一系列多碳糖之间复杂的生物转化后,5分子3-磷酸甘油醛先转化为1分子5-磷酸核酮糖,随之再生得到1分子RuBP.综上所述,每轮CBB循环固定1分子CO2,三轮循环后生成1分子3-磷酸甘油醛,共计消耗6分子NAD(P)H与9分子ATP.CBB循环依赖型微生物可以将甲酸作为唯一碳源和能源进行细胞生长.然而,CBB循环途径仍然存在一定的不足,如该循环是ATP利用效率最低的固碳途径之一[72].与甲酸直接同化相比,甲酸的氧化供能存在着能量浪费,因为电子从甲酸转移到氧化还原载体的过程需要消耗额外的能量[71].因此,CBB循环依赖型微生物的细胞生长速率与生产潜能仍然不够高,与实际的工业化生产仍然存在差距. ...
... [71].因此,CBB循环依赖型微生物的细胞生长速率与生产潜能仍然不够高,与实际的工业化生产仍然存在差距. ...
Thermodynamic constraints shape the structure of carbon fixation pathways
1
2012
... 还原性磷酸戊糖循环(reductive pentose-phosphate cycle),也称为Calvin-Benson-Bassham(CBB)循环,是唯一已知的能够利用甲酸进行微生物自养生长的天然碳同化途径[71].CBB循环主要包括三个过程[图3(a)]:首先是羧化作用,在关键酶核酮糖-1,5-二磷酸羧化酶/加氧酶的作用下,1分子CO2与1,5-二磷酸核酮糖(ribulose-1,5-bisphosphate,RuBP)分子进行反应,生成一个不稳定的六碳化合物,随之分解为2分子3-磷酸甘油酸;其次是还原作用,2分子3-磷酸甘油酸先转化为2分子1,3-二磷酸甘油酸,随之生成2分子3-磷酸甘油醛;最后是RuBP的再生,在经过一系列多碳糖之间复杂的生物转化后,5分子3-磷酸甘油醛先转化为1分子5-磷酸核酮糖,随之再生得到1分子RuBP.综上所述,每轮CBB循环固定1分子CO2,三轮循环后生成1分子3-磷酸甘油醛,共计消耗6分子NAD(P)H与9分子ATP.CBB循环依赖型微生物可以将甲酸作为唯一碳源和能源进行细胞生长.然而,CBB循环途径仍然存在一定的不足,如该循环是ATP利用效率最低的固碳途径之一[72].与甲酸直接同化相比,甲酸的氧化供能存在着能量浪费,因为电子从甲酸转移到氧化还原载体的过程需要消耗额外的能量[71].因此,CBB循环依赖型微生物的细胞生长速率与生产潜能仍然不够高,与实际的工业化生产仍然存在差距. ...
Adaptively evolved Escherichia coli for improved ability of formate utilization as a carbon source in sugar-free conditions
2
2019
... 丝氨酸循环(serine cycle)是多种甲基营养型微生物(如M. extorquens AM1[31])同化利用甲酸的代谢途径.该循环包含了二碳、三碳及四碳化合物之间的相互转化[图3(b)]:首先是四氢叶酸(tetrahydrofolic acid,简称THF)循环,即甲酸分子在甲酸四氢叶酸连接酶、次甲基四氢叶酸环水解酶和亚甲基四氢叶酸脱氢酶的先后作用下,生成5,10-亚甲基四氢叶酸,随后5,10-亚甲基四氢叶酸的甲基转移到二碳化合物甘氨酸上,生成三碳化合物丝氨酸;其次,丝氨酸依次转化为羟基丙酮酸、甘油酸、2-磷酸甘油酸与磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP),随后PEP经羧化作用固定1分子CO2生成四碳化合物草酰乙酸;然后,草酰乙酸依次转化为苹果酸与苹果酰辅酶A;最后,苹果酰辅酶A裂解为两个二碳化合物乙酰辅酶A与乙醛酸,其中乙醛酸能够再次转化为甘氨酸用于维持丝氨酸循环.综上所述,每轮丝氨酸循环能够同化1分子甲酸并固定1分子CO2,最终生成1分子乙酰辅酶A,共计消耗3分子NAD(P)H与3分子ATP.与甲酸氧化供能相比,丝氨酸循环可以同化甲酸并固定CO2,不仅避免了碳损失,而且还能在一定程度上减少能量浪费.通过在E. coli中引入异源的天然丝氨酸循环,同时结合实验室适应性进化策略,获得了能够同化利用甲酸合成乙醇的工程菌株[73],从而验证了丝氨酸循环在模式微生物中同化利用甲酸的可行性.然而,天然丝氨酸循环所涉及的酶和代谢反应比较多,且ATP利用效率仍然比较低,因此,利用该循环同化甲酸的微生物,目标代谢物的产量偏低,目前还不适合用于工业化生产. ...
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Beating the acetyl coenzyme A-pathway to the origin of life
1
2013
... 还原性乙酰辅酶A途径(reductive acetyl-CoA pathway),也称为Wood-Ljungdahl(WL)途径,是一种广泛存在于产乙酸菌、产甲烷菌与硫酸盐还原菌等厌氧微生物中的固碳途径.不同类型的厌氧微生物所具有的WL途径结构相似但有所差别,如产乙酸菌与产甲烷菌的WL途径存在着一碳载体、辅酶与路径酶等方面的差异[74].以产乙酸菌的WL途径为例[图3(c)],WL途径由甲基分支与羰基分支两条支路组成.在甲基分支中,甲酸到5,10-亚甲基四氢叶酸的部分与丝氨酸循环一致,随后5,10-亚甲基四氢叶酸先转化为5-甲基四氢叶酸,再实现甲基与钴铁硫蛋白(corrinoid ironsulfur protein,CoFeSP)间的连接,生成5-甲基钴铁硫蛋白;在羰基分支中,CO2在一氧化碳脱氢酶的作用下转化为CO;最后,甲基分支的产物5-甲基钴铁硫蛋白提供甲基,羰基分支的产物CO提供羰基,两者连接生成乙酰辅酶A.综上所述,WL途径能够同化1分子甲酸与1分子CO2,最终生成1分子乙酰辅酶A,共计消耗3分子NAD(P)H与1分子ATP.与丝氨酸循环相比,WL途径生成1分子乙酰辅酶A仅仅消耗了1分子ATP,且该途径不存在碳损失,是ATP利用效率最高的天然甲酸同化路径.然而,WL途径的蛋白质组学十分复杂,且包含对氧气非常敏感的路径酶,因此,该途径仅仅能够在严格厌氧的条件下运行,且代谢改造难度比较大[75-76],实现工业应用仍然存在挑战. ...
The formate bio-economy
1
2016
... 还原性乙酰辅酶A途径(reductive acetyl-CoA pathway),也称为Wood-Ljungdahl(WL)途径,是一种广泛存在于产乙酸菌、产甲烷菌与硫酸盐还原菌等厌氧微生物中的固碳途径.不同类型的厌氧微生物所具有的WL途径结构相似但有所差别,如产乙酸菌与产甲烷菌的WL途径存在着一碳载体、辅酶与路径酶等方面的差异[74].以产乙酸菌的WL途径为例[图3(c)],WL途径由甲基分支与羰基分支两条支路组成.在甲基分支中,甲酸到5,10-亚甲基四氢叶酸的部分与丝氨酸循环一致,随后5,10-亚甲基四氢叶酸先转化为5-甲基四氢叶酸,再实现甲基与钴铁硫蛋白(corrinoid ironsulfur protein,CoFeSP)间的连接,生成5-甲基钴铁硫蛋白;在羰基分支中,CO2在一氧化碳脱氢酶的作用下转化为CO;最后,甲基分支的产物5-甲基钴铁硫蛋白提供甲基,羰基分支的产物CO提供羰基,两者连接生成乙酰辅酶A.综上所述,WL途径能够同化1分子甲酸与1分子CO2,最终生成1分子乙酰辅酶A,共计消耗3分子NAD(P)H与1分子ATP.与丝氨酸循环相比,WL途径生成1分子乙酰辅酶A仅仅消耗了1分子ATP,且该途径不存在碳损失,是ATP利用效率最高的天然甲酸同化路径.然而,WL途径的蛋白质组学十分复杂,且包含对氧气非常敏感的路径酶,因此,该途径仅仅能够在严格厌氧的条件下运行,且代谢改造难度比较大[75-76],实现工业应用仍然存在挑战. ...
Third-generation biorefineries as the means to produce fuels and chemicals from CO2
1
2020
... 还原性乙酰辅酶A途径(reductive acetyl-CoA pathway),也称为Wood-Ljungdahl(WL)途径,是一种广泛存在于产乙酸菌、产甲烷菌与硫酸盐还原菌等厌氧微生物中的固碳途径.不同类型的厌氧微生物所具有的WL途径结构相似但有所差别,如产乙酸菌与产甲烷菌的WL途径存在着一碳载体、辅酶与路径酶等方面的差异[74].以产乙酸菌的WL途径为例[图3(c)],WL途径由甲基分支与羰基分支两条支路组成.在甲基分支中,甲酸到5,10-亚甲基四氢叶酸的部分与丝氨酸循环一致,随后5,10-亚甲基四氢叶酸先转化为5-甲基四氢叶酸,再实现甲基与钴铁硫蛋白(corrinoid ironsulfur protein,CoFeSP)间的连接,生成5-甲基钴铁硫蛋白;在羰基分支中,CO2在一氧化碳脱氢酶的作用下转化为CO;最后,甲基分支的产物5-甲基钴铁硫蛋白提供甲基,羰基分支的产物CO提供羰基,两者连接生成乙酰辅酶A.综上所述,WL途径能够同化1分子甲酸与1分子CO2,最终生成1分子乙酰辅酶A,共计消耗3分子NAD(P)H与1分子ATP.与丝氨酸循环相比,WL途径生成1分子乙酰辅酶A仅仅消耗了1分子ATP,且该途径不存在碳损失,是ATP利用效率最高的天然甲酸同化路径.然而,WL途径的蛋白质组学十分复杂,且包含对氧气非常敏感的路径酶,因此,该途径仅仅能够在严格厌氧的条件下运行,且代谢改造难度比较大[75-76],实现工业应用仍然存在挑战. ...
Functional cooperation of the glycine synthase-reductase and Wood-Ljungdahl pathways for autotrophic growth of Clostridium drakei
1
2020
... 还原性甘氨酸途径(reductive glycine pathway,简称为rGly途径),主要存在于硫酸盐还原菌(如D. desulfuricans)[46]和产乙酸菌(如梭状芽孢杆菌Clostridium drakei)[77]中.该途径主要包括三个模块[图3(d)]:首先,由甲酸到5,10-亚甲基四氢叶酸的转化;其次,5,10-亚甲基四氢叶酸在甘氨酸裂解系统(glycine cleavage system,GCS)的催化下,固定1分子CO2生成甘氨酸;最后,甘氨酸可通过两种路线转化为丙酮酸,一种为还原甘氨酸(reductive glycine,RG)路线,即5,10-亚甲基四氢叶酸的甲基转移到甘氨酸上生成丝氨酸,随后丝氨酸脱氨基形成丙酮酸,另一种为甘氨酸还原酶(glycine reductase,GR)路线,即甘氨酸依次转化为乙酰磷酸、乙酸与乙酰辅酶A,随后固定1分子CO2生成丙酮酸.综上所述,依赖于RG路线的rGly途径能够同化2分子甲酸和1分子CO2,最终生成1分子丙酮酸,共计消耗3分子NAD(P)H与2分子ATP;依赖于GR路线的rGly途径能够同化1分子甲酸和2分子CO2,最终生成1分子丙酮酸,共计消耗2分子NAD(P)H与1分子ATP.rGly途径是一种能够高效同化甲酸的代谢路径,具有许多优势:rGly途径对ATP的利用效率仅次于WL途径;rGly途径为线性途径,降低了代谢改造的难度,且多种微生物具有内源性的rGly途径[78];rGly途径与中心代谢途径的重叠较少,因此异源引入或改造内源rGly途径对本源代谢网络的干扰比较小;与丝氨酸循环和WL途径相比,rGly途径具有更高的代谢物灵活性,可以进一步转化为多种化学品.基于上述优势,rGly途径成为目前最具发展潜力的甲酸代谢路径,与此同时,对路径关键酶GCS的深入研究[79-80],有望进一步优化rGly途径.然而,对于具有天然rGly途径的微生物研究仍然较少,还不能满足现阶段甲酸生物经济的发展要求. ...
Recent progress in metabolic engineering of microbial formate assimilation
1
2020
... 还原性甘氨酸途径(reductive glycine pathway,简称为rGly途径),主要存在于硫酸盐还原菌(如D. desulfuricans)[46]和产乙酸菌(如梭状芽孢杆菌Clostridium drakei)[77]中.该途径主要包括三个模块[图3(d)]:首先,由甲酸到5,10-亚甲基四氢叶酸的转化;其次,5,10-亚甲基四氢叶酸在甘氨酸裂解系统(glycine cleavage system,GCS)的催化下,固定1分子CO2生成甘氨酸;最后,甘氨酸可通过两种路线转化为丙酮酸,一种为还原甘氨酸(reductive glycine,RG)路线,即5,10-亚甲基四氢叶酸的甲基转移到甘氨酸上生成丝氨酸,随后丝氨酸脱氨基形成丙酮酸,另一种为甘氨酸还原酶(glycine reductase,GR)路线,即甘氨酸依次转化为乙酰磷酸、乙酸与乙酰辅酶A,随后固定1分子CO2生成丙酮酸.综上所述,依赖于RG路线的rGly途径能够同化2分子甲酸和1分子CO2,最终生成1分子丙酮酸,共计消耗3分子NAD(P)H与2分子ATP;依赖于GR路线的rGly途径能够同化1分子甲酸和2分子CO2,最终生成1分子丙酮酸,共计消耗2分子NAD(P)H与1分子ATP.rGly途径是一种能够高效同化甲酸的代谢路径,具有许多优势:rGly途径对ATP的利用效率仅次于WL途径;rGly途径为线性途径,降低了代谢改造的难度,且多种微生物具有内源性的rGly途径[78];rGly途径与中心代谢途径的重叠较少,因此异源引入或改造内源rGly途径对本源代谢网络的干扰比较小;与丝氨酸循环和WL途径相比,rGly途径具有更高的代谢物灵活性,可以进一步转化为多种化学品.基于上述优势,rGly途径成为目前最具发展潜力的甲酸代谢路径,与此同时,对路径关键酶GCS的深入研究[79-80],有望进一步优化rGly途径.然而,对于具有天然rGly途径的微生物研究仍然较少,还不能满足现阶段甲酸生物经济的发展要求. ...
Structure-based dynamic analysis of the glycine cleavage system suggests key residues for control of a key reaction step
1
2020
... 还原性甘氨酸途径(reductive glycine pathway,简称为rGly途径),主要存在于硫酸盐还原菌(如D. desulfuricans)[46]和产乙酸菌(如梭状芽孢杆菌Clostridium drakei)[77]中.该途径主要包括三个模块[图3(d)]:首先,由甲酸到5,10-亚甲基四氢叶酸的转化;其次,5,10-亚甲基四氢叶酸在甘氨酸裂解系统(glycine cleavage system,GCS)的催化下,固定1分子CO2生成甘氨酸;最后,甘氨酸可通过两种路线转化为丙酮酸,一种为还原甘氨酸(reductive glycine,RG)路线,即5,10-亚甲基四氢叶酸的甲基转移到甘氨酸上生成丝氨酸,随后丝氨酸脱氨基形成丙酮酸,另一种为甘氨酸还原酶(glycine reductase,GR)路线,即甘氨酸依次转化为乙酰磷酸、乙酸与乙酰辅酶A,随后固定1分子CO2生成丙酮酸.综上所述,依赖于RG路线的rGly途径能够同化2分子甲酸和1分子CO2,最终生成1分子丙酮酸,共计消耗3分子NAD(P)H与2分子ATP;依赖于GR路线的rGly途径能够同化1分子甲酸和2分子CO2,最终生成1分子丙酮酸,共计消耗2分子NAD(P)H与1分子ATP.rGly途径是一种能够高效同化甲酸的代谢路径,具有许多优势:rGly途径对ATP的利用效率仅次于WL途径;rGly途径为线性途径,降低了代谢改造的难度,且多种微生物具有内源性的rGly途径[78];rGly途径与中心代谢途径的重叠较少,因此异源引入或改造内源rGly途径对本源代谢网络的干扰比较小;与丝氨酸循环和WL途径相比,rGly途径具有更高的代谢物灵活性,可以进一步转化为多种化学品.基于上述优势,rGly途径成为目前最具发展潜力的甲酸代谢路径,与此同时,对路径关键酶GCS的深入研究[79-80],有望进一步优化rGly途径.然而,对于具有天然rGly途径的微生物研究仍然较少,还不能满足现阶段甲酸生物经济的发展要求. ...
Improvement of glycine biosynthesis from one-carbon compounds and ammonia catalyzed by the glycine cleavage system in vitro
1
2022
... 还原性甘氨酸途径(reductive glycine pathway,简称为rGly途径),主要存在于硫酸盐还原菌(如D. desulfuricans)[46]和产乙酸菌(如梭状芽孢杆菌Clostridium drakei)[77]中.该途径主要包括三个模块[图3(d)]:首先,由甲酸到5,10-亚甲基四氢叶酸的转化;其次,5,10-亚甲基四氢叶酸在甘氨酸裂解系统(glycine cleavage system,GCS)的催化下,固定1分子CO2生成甘氨酸;最后,甘氨酸可通过两种路线转化为丙酮酸,一种为还原甘氨酸(reductive glycine,RG)路线,即5,10-亚甲基四氢叶酸的甲基转移到甘氨酸上生成丝氨酸,随后丝氨酸脱氨基形成丙酮酸,另一种为甘氨酸还原酶(glycine reductase,GR)路线,即甘氨酸依次转化为乙酰磷酸、乙酸与乙酰辅酶A,随后固定1分子CO2生成丙酮酸.综上所述,依赖于RG路线的rGly途径能够同化2分子甲酸和1分子CO2,最终生成1分子丙酮酸,共计消耗3分子NAD(P)H与2分子ATP;依赖于GR路线的rGly途径能够同化1分子甲酸和2分子CO2,最终生成1分子丙酮酸,共计消耗2分子NAD(P)H与1分子ATP.rGly途径是一种能够高效同化甲酸的代谢路径,具有许多优势:rGly途径对ATP的利用效率仅次于WL途径;rGly途径为线性途径,降低了代谢改造的难度,且多种微生物具有内源性的rGly途径[78];rGly途径与中心代谢途径的重叠较少,因此异源引入或改造内源rGly途径对本源代谢网络的干扰比较小;与丝氨酸循环和WL途径相比,rGly途径具有更高的代谢物灵活性,可以进一步转化为多种化学品.基于上述优势,rGly途径成为目前最具发展潜力的甲酸代谢路径,与此同时,对路径关键酶GCS的深入研究[79-80],有望进一步优化rGly途径.然而,对于具有天然rGly途径的微生物研究仍然较少,还不能满足现阶段甲酸生物经济的发展要求. ...
Escherichia coli in auto(trophic) mode
1
2019
... 通过在E. coli中重构卡尔文循环,首次实现了异养微生物向自养微生物的转化[57].通过敲除糖酵解途径中的磷酸果糖激酶和戊糖磷酸途径中的葡萄糖-6-磷酸脱氢酶,并异源表达核酮糖-1,5-二磷酸羧化酶、磷酸核酮糖激酶、碳酸酐酶和甲酸脱氢酶,实现了在E. coli中重构卡尔文循环[图4(a)].重构的卡尔文循环(reconstructed Calvin cycle)可以固定CO2合成生物量,同时氧化甲酸实现供能.在此基础上,将该循环与实验室适应性进化相结合,进化获得了自养型E. coli,这种基于甲酸供能的生物固碳模式在甲酸生物经济发展和未来工业生物制造方面具有极大的潜力[81].此外,随着近年来电化学法还原CO2生产甲酸[82-84]的深入研究,该自养系统中由于甲酸氧化速率高于固碳速率而导致的CO2净排放也有望得到优化与解决. ...
Recent advances in designing efficient electrocatalysts for electrochemical carbon dioxide reduction to formic acid/formate
1
2023
... 通过在E. coli中重构卡尔文循环,首次实现了异养微生物向自养微生物的转化[57].通过敲除糖酵解途径中的磷酸果糖激酶和戊糖磷酸途径中的葡萄糖-6-磷酸脱氢酶,并异源表达核酮糖-1,5-二磷酸羧化酶、磷酸核酮糖激酶、碳酸酐酶和甲酸脱氢酶,实现了在E. coli中重构卡尔文循环[图4(a)].重构的卡尔文循环(reconstructed Calvin cycle)可以固定CO2合成生物量,同时氧化甲酸实现供能.在此基础上,将该循环与实验室适应性进化相结合,进化获得了自养型E. coli,这种基于甲酸供能的生物固碳模式在甲酸生物经济发展和未来工业生物制造方面具有极大的潜力[81].此外,随着近年来电化学法还原CO2生产甲酸[82-84]的深入研究,该自养系统中由于甲酸氧化速率高于固碳速率而导致的CO2净排放也有望得到优化与解决. ...
Simultaneous anodic and cathodic formate production in a paired electrolyzer by CO2 reduction and glycerol oxidation
0
2023
Halogen-incorporated Sn catalysts for selective electrochemical CO2 reduction to formate
1
2023
... 通过在E. coli中重构卡尔文循环,首次实现了异养微生物向自养微生物的转化[57].通过敲除糖酵解途径中的磷酸果糖激酶和戊糖磷酸途径中的葡萄糖-6-磷酸脱氢酶,并异源表达核酮糖-1,5-二磷酸羧化酶、磷酸核酮糖激酶、碳酸酐酶和甲酸脱氢酶,实现了在E. coli中重构卡尔文循环[图4(a)].重构的卡尔文循环(reconstructed Calvin cycle)可以固定CO2合成生物量,同时氧化甲酸实现供能.在此基础上,将该循环与实验室适应性进化相结合,进化获得了自养型E. coli,这种基于甲酸供能的生物固碳模式在甲酸生物经济发展和未来工业生物制造方面具有极大的潜力[81].此外,随着近年来电化学法还原CO2生产甲酸[82-84]的深入研究,该自养系统中由于甲酸氧化速率高于固碳速率而导致的CO2净排放也有望得到优化与解决. ...
Formate assimilation: the metabolic architecture of natural and synthetic pathways
1
2016
... 基于丝氨酸循环,在E. coli中构建了高丝氨酸循环(homoserine cycle)[59][图4(c)].首先,甲酸在乙酰辅酶A合成酶和乙醛脱氢酶的作用下转化为甲醛,甲醛作为一种高活性化合物,更易于被同化进入代谢系统[56,85];其次,在丝氨酸醛缩酶的作用下,甲醛与甘氨酸反应生成丝氨酸,随之丝氨酸脱氨形成丙酮酸;然后,另一分子甲醛与丙酮酸在4-羟基-2-氧代丁酸(4-hydroxy-2-oxobutanoate,HOB)醛缩酶的作用下,生成非天然代谢物HOB;随后,氨基被结合到HOB上生成高丝氨酸,高丝氨酸在高丝氨酸激酶与苏氨酸合成酶的作用下转化为苏氨酸;最后,在苏氨酸醛缩酶的催化下,苏氨酸裂解为甘氨酸与乙醛.其中,甘氨酸重新进入循环,而乙醛分子经乙醛脱氢酶的催化,生成乙酰辅酶A并输出循环.综上所述,每轮高丝氨酸循环能够同化2分子甲酸,最终生成1分子乙酰辅酶A,共计消耗1分子NADH与3分子ATP.高丝氨酸循环是在改良丝氨酸循环的基础上,对天然丝氨酸循环进行的更深层的优化,具有明显的优势:以甲醛替代CO2进入同化路径,改善了羧化反应所造成的限速作用,减少了还原力的使用,有利于支持更高的生物量产量;该循环路径所涉及的酶均为E. coli的内源酶,保证了酶的催化活性;相比于丝氨酸循环,该循环所包含的酶促反应较少,且与中心代谢途径的重叠也较少[59].然而,甲醛与高丝氨酸对E. coli都具有一定的毒性作用.因此,深入探究E. coli的毒性机理与解毒机制[86-88],对于进一步优化高丝氨酸循环具有重要意义. ...
Formaldehyde stress responses in bacterial pathogens
1
2016
... 基于丝氨酸循环,在E. coli中构建了高丝氨酸循环(homoserine cycle)[59][图4(c)].首先,甲酸在乙酰辅酶A合成酶和乙醛脱氢酶的作用下转化为甲醛,甲醛作为一种高活性化合物,更易于被同化进入代谢系统[56,85];其次,在丝氨酸醛缩酶的作用下,甲醛与甘氨酸反应生成丝氨酸,随之丝氨酸脱氨形成丙酮酸;然后,另一分子甲醛与丙酮酸在4-羟基-2-氧代丁酸(4-hydroxy-2-oxobutanoate,HOB)醛缩酶的作用下,生成非天然代谢物HOB;随后,氨基被结合到HOB上生成高丝氨酸,高丝氨酸在高丝氨酸激酶与苏氨酸合成酶的作用下转化为苏氨酸;最后,在苏氨酸醛缩酶的催化下,苏氨酸裂解为甘氨酸与乙醛.其中,甘氨酸重新进入循环,而乙醛分子经乙醛脱氢酶的催化,生成乙酰辅酶A并输出循环.综上所述,每轮高丝氨酸循环能够同化2分子甲酸,最终生成1分子乙酰辅酶A,共计消耗1分子NADH与3分子ATP.高丝氨酸循环是在改良丝氨酸循环的基础上,对天然丝氨酸循环进行的更深层的优化,具有明显的优势:以甲醛替代CO2进入同化路径,改善了羧化反应所造成的限速作用,减少了还原力的使用,有利于支持更高的生物量产量;该循环路径所涉及的酶均为E. coli的内源酶,保证了酶的催化活性;相比于丝氨酸循环,该循环所包含的酶促反应较少,且与中心代谢途径的重叠也较少[59].然而,甲醛与高丝氨酸对E. coli都具有一定的毒性作用.因此,深入探究E. coli的毒性机理与解毒机制[86-88],对于进一步优化高丝氨酸循环具有重要意义. ...
Toxic effect and inability of L-homoserine to be a nitrogen source for growth of Escherichia coli resolved by a combination of in vivo evolution engineering and omics analyses
0
2022
Unravelling formaldehyde metabolism in bacteria: road towards synthetic methylotrophy
1
2022
... 基于丝氨酸循环,在E. coli中构建了高丝氨酸循环(homoserine cycle)[59][图4(c)].首先,甲酸在乙酰辅酶A合成酶和乙醛脱氢酶的作用下转化为甲醛,甲醛作为一种高活性化合物,更易于被同化进入代谢系统[56,85];其次,在丝氨酸醛缩酶的作用下,甲醛与甘氨酸反应生成丝氨酸,随之丝氨酸脱氨形成丙酮酸;然后,另一分子甲醛与丙酮酸在4-羟基-2-氧代丁酸(4-hydroxy-2-oxobutanoate,HOB)醛缩酶的作用下,生成非天然代谢物HOB;随后,氨基被结合到HOB上生成高丝氨酸,高丝氨酸在高丝氨酸激酶与苏氨酸合成酶的作用下转化为苏氨酸;最后,在苏氨酸醛缩酶的催化下,苏氨酸裂解为甘氨酸与乙醛.其中,甘氨酸重新进入循环,而乙醛分子经乙醛脱氢酶的催化,生成乙酰辅酶A并输出循环.综上所述,每轮高丝氨酸循环能够同化2分子甲酸,最终生成1分子乙酰辅酶A,共计消耗1分子NADH与3分子ATP.高丝氨酸循环是在改良丝氨酸循环的基础上,对天然丝氨酸循环进行的更深层的优化,具有明显的优势:以甲醛替代CO2进入同化路径,改善了羧化反应所造成的限速作用,减少了还原力的使用,有利于支持更高的生物量产量;该循环路径所涉及的酶均为E. coli的内源酶,保证了酶的催化活性;相比于丝氨酸循环,该循环所包含的酶促反应较少,且与中心代谢途径的重叠也较少[59].然而,甲醛与高丝氨酸对E. coli都具有一定的毒性作用.因此,深入探究E. coli的毒性机理与解毒机制[86-88],对于进一步优化高丝氨酸循环具有重要意义. ...
Introduction of glycine synthase enables uptake of exogenous formate and strongly impacts the metabolism in Clostridium pasteurianum
1
2021
... Ftl、Fch、Mtd、GlyA、Sda(内源酶)
[89] | | Pseudomonas putida | Ftl、Fch、Mtd(源自Methylobacterium extorquens AM1) ...
Novel computational and experimental approaches for investigating the thermodynamics of metabolic networks
1
2022
... 随着计算机辅助路径设计[90]与酶工程[91-92]等技术的发展,研究人员在开发新型甲酸利用路径(图5)时不再局限于自然界已有的酶促反应,这不仅有利于简化甲酸利用路径,而且还能够拓展甲酸营养型微生物的改造空间. ...
Engineering new catalytic activities in enzymes
1
2020
... 随着计算机辅助路径设计[90]与酶工程[91-92]等技术的发展,研究人员在开发新型甲酸利用路径(图5)时不再局限于自然界已有的酶促反应,这不仅有利于简化甲酸利用路径,而且还能够拓展甲酸营养型微生物的改造空间. ...
Enzyme activity engineering based on sequence co-evolution analysis
1
2022
... 随着计算机辅助路径设计[90]与酶工程[91-92]等技术的发展,研究人员在开发新型甲酸利用路径(图5)时不再局限于自然界已有的酶促反应,这不仅有利于简化甲酸利用路径,而且还能够拓展甲酸营养型微生物的改造空间. ...
Computational protein design enables a novel one-carbon assimilation pathway
1
2015
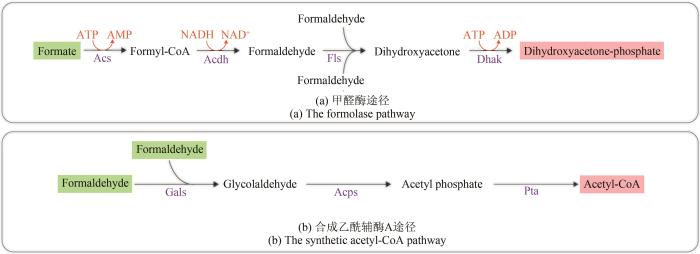
... 以荧光假单胞菌(Pseudomonas fluorescens)的苯甲醛裂解酶为基础,设计并优化获得了一种新酶:甲醛酶(formolase)[93].在此基础上,重构了一条新型甲酸同化路径,即甲醛酶途径(formolase pathway),该途径由四步酶促反应组成[图5(a)]:首先,甲酸在乙酰辅酶A合成酶与乙醛脱氢酶的作用下转化为甲醛;其次,在甲醛酶的催化作用下,3分子甲醛缩合生成1分子二羟基丙酮;最后,二羟基丙酮在二羟基丙酮激酶的作用下转化为微生物的中心代谢物磷酸二羟丙酮.综上所述,该路径能够同化3分子甲酸生成1分子磷酸二羟丙酮,共计消耗3分子NADH与4分子ATP.甲醛酶路径为线性反应路径,仅需4种酶即可完成路径催化,然而,甲醛酶的酶促反应效率比较低,从而限制了该路径的应用潜能.通过酶工程策略对甲醛酶途径的甲醛酶与乙醛脱氢酶进行改造,能够有效提高该路径对甲酸的利用效率[94-95]. ...
Light-driven CO2 sequestration in Escherichia coli to achieve theoretical yield of chemicals
1
2021
... 以荧光假单胞菌(Pseudomonas fluorescens)的苯甲醛裂解酶为基础,设计并优化获得了一种新酶:甲醛酶(formolase)[93].在此基础上,重构了一条新型甲酸同化路径,即甲醛酶途径(formolase pathway),该途径由四步酶促反应组成[图5(a)]:首先,甲酸在乙酰辅酶A合成酶与乙醛脱氢酶的作用下转化为甲醛;其次,在甲醛酶的催化作用下,3分子甲醛缩合生成1分子二羟基丙酮;最后,二羟基丙酮在二羟基丙酮激酶的作用下转化为微生物的中心代谢物磷酸二羟丙酮.综上所述,该路径能够同化3分子甲酸生成1分子磷酸二羟丙酮,共计消耗3分子NADH与4分子ATP.甲醛酶路径为线性反应路径,仅需4种酶即可完成路径催化,然而,甲醛酶的酶促反应效率比较低,从而限制了该路径的应用潜能.通过酶工程策略对甲醛酶途径的甲醛酶与乙醛脱氢酶进行改造,能够有效提高该路径对甲酸的利用效率[94-95]. ...
Synergistic metabolism of glucose and formate increases the yield of short-chain organic acids in Escherichia coli
2
2022
... 以荧光假单胞菌(Pseudomonas fluorescens)的苯甲醛裂解酶为基础,设计并优化获得了一种新酶:甲醛酶(formolase)[93].在此基础上,重构了一条新型甲酸同化路径,即甲醛酶途径(formolase pathway),该途径由四步酶促反应组成[图5(a)]:首先,甲酸在乙酰辅酶A合成酶与乙醛脱氢酶的作用下转化为甲醛;其次,在甲醛酶的催化作用下,3分子甲醛缩合生成1分子二羟基丙酮;最后,二羟基丙酮在二羟基丙酮激酶的作用下转化为微生物的中心代谢物磷酸二羟丙酮.综上所述,该路径能够同化3分子甲酸生成1分子磷酸二羟丙酮,共计消耗3分子NADH与4分子ATP.甲醛酶路径为线性反应路径,仅需4种酶即可完成路径催化,然而,甲醛酶的酶促反应效率比较低,从而限制了该路径的应用潜能.通过酶工程策略对甲醛酶途径的甲醛酶与乙醛脱氢酶进行改造,能够有效提高该路径对甲酸的利用效率[94-95]. ...
... 利用甲酸与其他碳源对微生物进行共底物培养也是实现甲酸有效利用的方法之一,如甲酸与其他一碳化合物(如甲醇和甲醛等)可以实现协同代谢.在甲酸与甲醇的协同代谢过程中,甲醇脱氢酶能够催化甲醇转化为甲醛,而甲醛经甲醛脱氢酶的催化可进一步转化为甲酸,上述过程均能够产生还原力,从而为甲酸营养型微生物的生长提供更多能量.然而,传统碳源(如葡萄糖和蔗糖等)与甲酸的协同代谢具有更大的优势,不仅有利于传统生物经济向甲酸生物经济的过渡,而且更加适用于现阶段的工业应用[图7(c)].曼氏产琥珀酸菌(Mannheimia succiniciproducens)是公认的高效生产琥珀酸的菌种之一,以阻断副产物合成途径的M. succiniciproducens为底盘微生物,通过过表达源自M. extorquens的甲酸脱氢酶后,实现了工程菌株协同代谢葡萄糖(或蔗糖)和甲酸,使得琥珀酸得率达到了1.41 mol/mol,接近于琥珀酸的最大理论得率1.5 mol/mol[98].另外,传统碳源与甲酸的协同代谢也拓展了模式微生物(如E. coli)的工业应用范围.例如,通过在E. coli中构建能够协同代谢葡萄糖和甲酸的合成途径SMGF,使得工程菌株由葡萄糖合成丙酮酸的得率达到了理论糖酵解得率的94%(达到1.88 mol/mol)[95]. ...
Synthetic methanol and formate assimilation via modular engineering and selection strategies
1
2019
... 路径模块化优化作为一种理性的代谢工程策略,通过将甲酸同化路径划分为几个代谢模块,并利用相应的营养缺陷型菌株测试各个模块的活性与限制性瓶颈,通过解除瓶颈最终实现甲酸同化路径的整体优化与适配[96][图6(e)].例如,借助模块化工程策略,通过组合rTHF模块与丝氨酸-苏氨酸循环,验证了甲酸同化在E. coli中的代谢可行性[60].首先,通过在一碳与甘氨酸营养缺陷型菌株中过表达异源Ftl,甲酸能够回补菌株的一碳营养缺陷,从而证明了一碳模块的活性;其次,通过在丝氨酸营养缺陷型菌株中过表达rTHF模块的Fch/ Mtd双功能酶(FolD)与GlyA,工程菌株实现了在甲酸与甘氨酸上进行生长;最后,通过在丝氨酸营养缺陷型菌株中引入丝氨酸-苏氨酸循环,使工程菌株能够在甲酸与葡萄糖上进行细胞生长,不再需要提供外源甘氨酸.上述研究为后续甲酸同化路径的构建提供了重要参考,模块化工程不仅实现了甲酸营养型E. coli的构建[17],而且还完成了P. putida甲酸代谢改造[70]等,从而拓展了同化甲酸的底盘微生物. ...
Adaptive laboratory evolution—principles and applications for biotechnology
1
2013
... 实验室适应性进化(adaptive laboratory evolution,ALE)是工业微生物领域常用的方法之一,能够在实验室条件下模拟自然进化对有益的遗传突变进行富集[97][图7(a)].与理性代谢工程策略相比,ALE无需对微生物复杂的代谢系统进行改造,不仅在一定程度上减少了工作量,而且还能够绕开现阶段代谢工程改造的理论与技术限制(尤其是非模式微生物).因此,ALE在甲酸营养型微生物的构建与应用方面具有明显优势,主要包括两种进化方式:①ALE用于推动甲酸利用路径在代谢网络中的运行,在提高路径代谢流通量的同时改善菌株的细胞生长[57,64].例如,在E. coli中引入异源Ftl(来自克氏梭菌Clostridium kluyveri)后,采用Turbidostat培养方法对菌株进行定向进化,提高了重构rTHF-rgcv途径在代谢网络中的代谢流通量,最终获得的菌株E. coli G4670和E. coli G4671能够利用甲酸和CO2合成甘氨酸和丝氨酸[64].在此基础上,通过进一步的长期进化,获得的菌株E. coli G5222和E. coli G5225能够利用甲酸与CO2为唯一碳源进行细胞生长[65].②ALE用于提高菌株在甲酸上的细胞生长与代谢能力,主要针对经过一系列代谢改造后具有一定甲酸代谢能力的工程菌株[16-17,70,73],或者本身不经改造即可天然利用甲酸的菌株(如C. necator H16[36]).例如,通过在E. coli中构建rTHF-rgcv途径与能量再生模块,实现了菌株利用甲酸为唯一碳源和能源进行细胞生长.随后,在补料-分批模式下对菌株进行短期进化,在13个进化周期内,菌株的倍增时间缩短至不到8 h,生物量产量提高至2.3 g CDW/mol甲酸[17].在此基础上,采用类似的进化方式,将菌株的倍增时间进一步缩短至6 h,生物量产量提高至3.3 g CDW/mol甲酸,同时也提高了菌株对甲酸的耐受性[66]. ...
Formic acid as a secondary substrate for succinic acid production by metabolically engineered Mannheimia succiniciproducens
1
2017
... 利用甲酸与其他碳源对微生物进行共底物培养也是实现甲酸有效利用的方法之一,如甲酸与其他一碳化合物(如甲醇和甲醛等)可以实现协同代谢.在甲酸与甲醇的协同代谢过程中,甲醇脱氢酶能够催化甲醇转化为甲醛,而甲醛经甲醛脱氢酶的催化可进一步转化为甲酸,上述过程均能够产生还原力,从而为甲酸营养型微生物的生长提供更多能量.然而,传统碳源(如葡萄糖和蔗糖等)与甲酸的协同代谢具有更大的优势,不仅有利于传统生物经济向甲酸生物经济的过渡,而且更加适用于现阶段的工业应用[图7(c)].曼氏产琥珀酸菌(Mannheimia succiniciproducens)是公认的高效生产琥珀酸的菌种之一,以阻断副产物合成途径的M. succiniciproducens为底盘微生物,通过过表达源自M. extorquens的甲酸脱氢酶后,实现了工程菌株协同代谢葡萄糖(或蔗糖)和甲酸,使得琥珀酸得率达到了1.41 mol/mol,接近于琥珀酸的最大理论得率1.5 mol/mol[98].另外,传统碳源与甲酸的协同代谢也拓展了模式微生物(如E. coli)的工业应用范围.例如,通过在E. coli中构建能够协同代谢葡萄糖和甲酸的合成途径SMGF,使得工程菌株由葡萄糖合成丙酮酸的得率达到了理论糖酵解得率的94%(达到1.88 mol/mol)[95]. ...
Microbiological surveillance of biogas plants: targeting acetogenic community
1
2021
... 在甲酸利用的微生物方面,深入探究天然甲酸利用微生物的甲酸代谢机制,针对微生物的生化特征与代谢特性,开发相应的分子遗传学操作工具与检测技术,如高通量测序技术[99]与同位素标记检测技术[100]等,从而拓展天然甲酸利用微生物的应用空间.目前,模式微生物利用甲酸的研究主要聚焦于E. coli,需要进一步拓展至改造与提高其他模式微生物(如S. cerevisiae与P. pastoris等)的甲酸代谢能力.此外,通过挖掘与开发新型甲酸利用微生物,也能够进一步拓宽甲酸利用微生物底盘与代谢产物范围. ...
Novel quantitative method for individual isotopomer of organic acids from 13C tracer experiments determines carbon flow in acetogenesis
1
2023
... 在甲酸利用的微生物方面,深入探究天然甲酸利用微生物的甲酸代谢机制,针对微生物的生化特征与代谢特性,开发相应的分子遗传学操作工具与检测技术,如高通量测序技术[99]与同位素标记检测技术[100]等,从而拓展天然甲酸利用微生物的应用空间.目前,模式微生物利用甲酸的研究主要聚焦于E. coli,需要进一步拓展至改造与提高其他模式微生物(如S. cerevisiae与P. pastoris等)的甲酸代谢能力.此外,通过挖掘与开发新型甲酸利用微生物,也能够进一步拓宽甲酸利用微生物底盘与代谢产物范围. ...
Toward a glycyl radical enzyme containing synthetic bacterial microcompartment to produce pyruvate from formate and acetate
1
2022
... 在甲酸利用的代谢路径方面,结合新型的基因编辑工具与路径酶改造技术,进一步优化现有的甲酸利用路径与路径酶.结合生物信息学与计算机算法,进一步设计与构建更加简单高效的甲酸利用路径.另外,通过构建微生物细胞微区室等方式,提高氧气敏感性路径酶在甲酸同化过程中的可实用性.在E. coli中设计能够封存丙酮酸甲酸裂解酶和磷酸酰基转移酶的微区室,可以有效地将甲酸和乙酰磷酸转化为丙酮酸[101].这类微生物细胞微区室的构建,不仅能够降低其他路径对甲酸同化路径的代谢干扰,而且对氧气敏感性路径酶在需氧生物中的应用具有重要的借鉴意义. ...
Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs
1
2013
... 在甲酸利用的代谢工程策略方面,开发更有效的代谢工程改造策略,优化微生物的甲酸代谢网络.通过探究sRNA对微生物基因表达的微调机制,并将该机制应用于微生物的甲酸代谢网络改造,有利于进一步优化相关基因在甲酸同化过程中的表达水平,从而提高甲酸营养型微生物的甲酸同化效率[102-103].此外,通过优化甲酸脱氢酶的催化性能、建立能够评估辅因子再生系统的微生物体内平台等策略,进一步完善甲酸代谢的能量再生机制.例如,通过在E. coli中敲除二氢硫辛酸脱氢酶基因,消除了丙酮酸脱氢酶和α-酮戊二酸脱氢酶的活性,从而构建了NADH和ATP的营养缺陷型菌株,并用于评估NAD+依赖型甲酸脱氢酶和甲醇脱氢酶,为甲酸代谢的能量机制评估提供了一种有效的策略[104].在此基础上,进一步采用实验室适应性进化等策略,提高甲酸利用微生物的工业化生产潜能. ...
An overview of gene regulation in bacteria by small RNAs derived from mRNA 3′ ends
1
2022
... 在甲酸利用的代谢工程策略方面,开发更有效的代谢工程改造策略,优化微生物的甲酸代谢网络.通过探究sRNA对微生物基因表达的微调机制,并将该机制应用于微生物的甲酸代谢网络改造,有利于进一步优化相关基因在甲酸同化过程中的表达水平,从而提高甲酸营养型微生物的甲酸同化效率[102-103].此外,通过优化甲酸脱氢酶的催化性能、建立能够评估辅因子再生系统的微生物体内平台等策略,进一步完善甲酸代谢的能量再生机制.例如,通过在E. coli中敲除二氢硫辛酸脱氢酶基因,消除了丙酮酸脱氢酶和α-酮戊二酸脱氢酶的活性,从而构建了NADH和ATP的营养缺陷型菌株,并用于评估NAD+依赖型甲酸脱氢酶和甲醇脱氢酶,为甲酸代谢的能量机制评估提供了一种有效的策略[104].在此基础上,进一步采用实验室适应性进化等策略,提高甲酸利用微生物的工业化生产潜能. ...
An "energy-auxotroph" Escherichia coli provides an in vivo platform for assessing NADH regeneration systems
1
2020
... 在甲酸利用的代谢工程策略方面,开发更有效的代谢工程改造策略,优化微生物的甲酸代谢网络.通过探究sRNA对微生物基因表达的微调机制,并将该机制应用于微生物的甲酸代谢网络改造,有利于进一步优化相关基因在甲酸同化过程中的表达水平,从而提高甲酸营养型微生物的甲酸同化效率[102-103].此外,通过优化甲酸脱氢酶的催化性能、建立能够评估辅因子再生系统的微生物体内平台等策略,进一步完善甲酸代谢的能量再生机制.例如,通过在E. coli中敲除二氢硫辛酸脱氢酶基因,消除了丙酮酸脱氢酶和α-酮戊二酸脱氢酶的活性,从而构建了NADH和ATP的营养缺陷型菌株,并用于评估NAD+依赖型甲酸脱氢酶和甲醇脱氢酶,为甲酸代谢的能量机制评估提供了一种有效的策略[104].在此基础上,进一步采用实验室适应性进化等策略,提高甲酸利用微生物的工业化生产潜能. ...
/
|
〈
|
![]()
|
〉
|
|