|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
DeepSeek model analysis and its applications in AI-assistant protein engineering
Synthetic Biology Journal
2025, 6 (3):
636-650.
DOI: 10.12211/2096-8280.2025-041
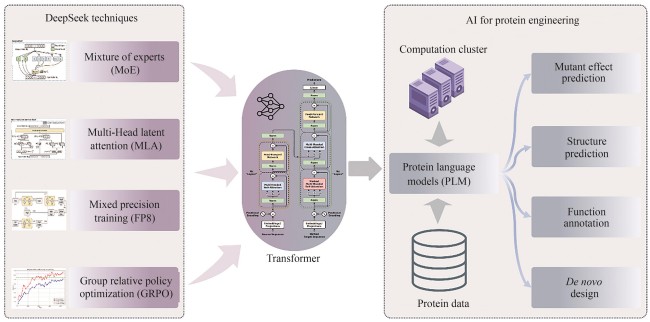
In early 2025, Hangzhou DeepSeek AI Foundation Technology Research Co., Ltd. released and open-sourced its independently developed DeepSeek-R1 conversational large language model. This model exhibits extremely low inference costs and outstanding chain-of-thought reasoning capabilities, performing comparably to, and in some tasks surpassing, proprietary models like GPT-4o and o1. This achievement has garnered significant international attention. Furthermore, DeepSeek’s excellent performance in Chinese conversations and its free-for-commercial-use strategy have ignited a wave of deployment and application within China, thereby promoting the widespread adoption and development of AI technology. This work systematically analyzes the architectural design, training methodology, and inference mechanisms of the DeepSeek model, exploring the transfer potential and application prospects of its core technologies in AI-assistant protein research. The DeepSeek model integrates several cutting-edge, independently innovated technologies, including a multi-head latent attention mechanism, mixture-of-experts (MoE) with load balancing, and low-precision training. These innovations have substantially reduced the training and inference costs for Transformer models. Although DeepSeek was originally designed for human language understanding and generation, its optimization techniques hold significant reference value for pre-trained language models with proteins, which are also based on the Transformer architecture. By leveraging the key technologies employed in DeepSeek, protein language models are expected to achieve substantial reductions in training and inference costs.
Table 2
Protein language models enhanced by introducing external knowledge
Extracts from the Article
人工智能,尤其是深度学习与大模型的发展,正在深刻变革科学研究的范式。2024年诺贝尔化学奖和物理学奖分别颁发给了人工智能预测蛋白质结构预测与神经网络,标志着人工智能已成为推动基础科学进步的重要技术[1]。在生物工程领域,蛋白质的理解与设计是核心研究方向之一。近年来,受自然语言处理中预训练语言模型的启发,蛋白质预训练语言模型应运而生。该类模型通过对海量未标注蛋白质序列数据进行自监督学习,能够有效捕捉序列中的“语法”与“语义”特征,从而获得高质量的蛋白质表示,并可应用于结构预测、功能注释等下游任务,甚至具备生成全新蛋白质序列的能力[2]。当前主流的蛋白质预训练模型多基于深度自注意力变换网络[3](Transformer),在模型设计与技术路径上与自然语言处理领域存在着高度的一致性。因此,自然语言模型研究领域针对Transformer所提出的各类优化策略,如注意力机制的改进、高效微调方法等,均可较为便捷地迁移至蛋白质语言模型的研究中。
ESM-2是由Meta公司Lin等[19]提出的一种基于掩码语言建模(masked language modeling, MLM)的蛋白质语言模型,专注于蛋白质表示学习与结构预测等下游任务,是目前应用广泛的AI蛋白质表示模型之一。作为典型的Transformer 编码器架构,ESM-2基于双向自注意力机制构建,最大版本参数量约为150亿。由于其推理过程采用并行解码方式,能够一次性完成整个蛋白质序列的编码,无需依赖KV-Cache等机制,因此DeepSeek提出的多头潜在注意力技术在该模型中的应用空间较为有限。混合专家网络及其负载均衡策略仍可在大规模特征提取场景下发挥作用,通过引入稀疏激活机制降低推理成本,但是这需要对模型进行重新训练。此外,FP8低精度训练作为一种基础性计算优化手段,可直接应用于ESM-2,有效降低计算资源消耗。
总体而言,DeepSeek所提出的模型优化技术在蛋白质语言模型中的应用价值与其具体用途相关。对于以表征学习为主的模型(如ESM-2),其优化空间相对有限;而对于以生成任务为核心的模型(如ProGEN),这些技术展现出较高的实用价值,特别是在需要生成大量候选蛋白质序列的场景下,能够在不牺牲模型性能的前提下显著降低计算成本,具有工程应用价值。
蛋白质语言模型生成或编码一条序列所需的计算机运行成本低于实验室中实现表达、纯化与功能检测的成本。因此,在当前AI蛋白质设计流程中,模型推理成本在整个研发链条中的占比较低,似乎表明假设2同样不成立,限制了DeepSeek提出的推理优化技术的直接应用空间。然而,随着模型即服务[45](model-as-a-service, MaaS)模式在蛋白质建模领域的推广,DeepSeek的优化技术能够发挥较大的作用,特别是在高并发、多用户使用场景下,推理效率的提升将有助于显著降低服务器运行与维护成本。
外部知识整合进模型的方式主要包括通过多任务学习作为学习目标(方式1)、作为模型额外的输入(方式2)两种。其中方式1将多任务学习作为学习目标的优势在于,仅需要在训练模型时提供标签即可,在推理时无需提供标签。因此,大多数基于功能属性注释增强的蛋白质语言模型均属于方式1,因为推理的场景下序列不一定会有标签。而方式2的优势在于引入的方式更为直接,在推理时模型能够高效利用推理数据的外部知识。因此,结构和MSA这种能够通过AlphaFold预测和序列比对软件获取的知识大多数可以作为额外的整合进入模型,以达到更好的模型性能。表2列举了一些目前常见的引入外部知识增强的蛋白质语言模型。
在构建高质量的数据集方面,Fournier等[49]提出,参数量大的模型不一定表现得好,开发更好的蛋白质语言模型除了扩大参数规模外,还可以通过构造高质量的数据来提升蛋白质语言模型。其开发的AMPLIFY模型的预训练数据集来源于UniRef100、SCOP与OAS[67]等多个数据库,能够提升蛋白质语言模型的表示能力,其提出,在训练模型时使用降重的方法对性能有害。在参数量大小远小于ESM-2的情况下,其模型完成了对ESM-2性能的超越。
在内部架构方面,可以从应用扩展定律、构建高质量数据集与使用非Transformer架构三个方面来改进蛋白质语言模型. ... Observed antibody space: a diverse database of cleaned, annotated, and translated unpaired and paired antibody sequences 1 2022 ... 在构建高质量的数据集方面,Fournier等[ Sequence modeling and design from molecular to genome scale with Evo 1 2024 ... 在非Transformer架构方面,研究者针对超长序列的问题,提出了多种架构.例如,Nguyen等提出了Evo模型[ Deep generative models of genetic variation capture the effects of mutations 1 2018 ... 蛋白质语言模型作为AI蛋白质设计领域的重要工具,正逐步展现出其在多种应用场景中的核心价值.在蛋白质功能注释方面,这类模型通过学习序列的深层隐空间表征,有效摆脱了对传统手工特征工程的依赖,其所生成的表示能够捕捉序列中蕴含的潜在生物学信息,并在多项功能预测与注释任务中展现出显著优势.在蛋白质及酶的工程改造中,蛋白质语言模型借助大规模预训练,掌握了自然序列的分布规律,从而理解进化过程中所体现的选择偏好[ Entropy-driven zero-shot deep learning model selection for viral proteins 1 2025 ... 蛋白质语言模型作为AI蛋白质设计领域的重要工具,正逐步展现出其在多种应用场景中的核心价值.在蛋白质功能注释方面,这类模型通过学习序列的深层隐空间表征,有效摆脱了对传统手工特征工程的依赖,其所生成的表示能够捕捉序列中蕴含的潜在生物学信息,并在多项功能预测与注释任务中展现出显著优势.在蛋白质及酶的工程改造中,蛋白质语言模型借助大规模预训练,掌握了自然序列的分布规律,从而理解进化过程中所体现的选择偏好[ /
Other Images/Table from this Article
|