|
||
|
CRISPR/Cas systems and their applications in gene editing with filamentous fungi
Synthetic Biology Journal
2024, 5 (3):
672-693.
DOI: 10.12211/2096-8280.2023-097
Filamentous fungi, which present distinct morphology and cell structure, play a critical role in human health as well as industrial and agricultural production. However, the unique characteristics of filamentous fungi make them difficult to be manipulated with traditional genetic engineering methods. Thus, the development of an efficient gene editing system is essential for exploring biological resources and understanding metabolic processes in filamentous fungi. The development of the Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR associated protein (CRISPR/Cas) system promotes more efficient and effective gene editing in different species, and brings a revolutionary breakthrough in fungal fundamental research and applications. In this review, we first briefly introduce the history, working mechanism, and classifications of the CRISPR/Cas mediated gene editing system. Next, we comment the functional components of CRISPR/Cas9 such as selective marker, Cas9 and gRNA and the delivery methods of these components in various filamentous fungi. Furthermore, we systematically discuss the applications of CRISPR related technologies, including CRISPR/Cas12, base-editor, CRISPRa, CRISPRi and CRISPR mediated epigenetic regulation, in the genetic engineering of filamentous fungi, particularly in marine-derived filamentous fungi. Finally, we address challenges with relative low gene editing efficiency and off-targets effects in engineering filamentous fungi, and highlight the potential solutions for developing novel CRISPR/Cas-based gene editing systems. This review can provide guidance for developing an efficient gene editing platform in filamentous fungi and pave the way for further exploration of the secondary metabolites and establishment of robust fungal cell factories.

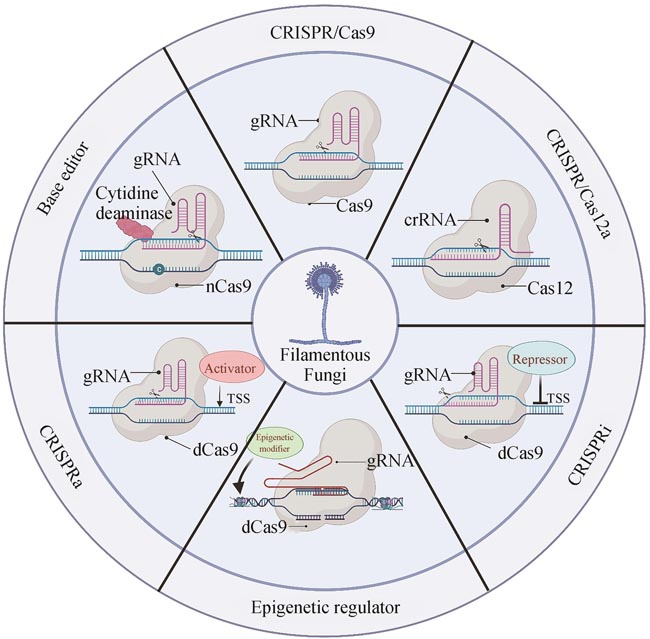
Fig. 3
Applications of next-generation CRISPR technologies in engineering filamentous fungi
Extracts from the Article
自2012年CRISPR/Cas技术开发以来,科学家们不断地为CRISPR工具箱添砖加瓦,目前以CRISPR/Cas为基础开发的多个基因编辑/调控技术,如CRISPR/Cas12a、CRISPR单碱基编辑器(CRISPR base editor)、CRISPR基因调控技术等都在丝状真菌中得以应用(图3),推动并加速了丝状真菌的遗传改造和基因功能解析。
CRISPR/Cas除了可用上述的基因编辑功能来灭活各种调控因子或替换启动子进而影响基因的表达,还可将丧失切割活性但仍具有RNA引导下与DNA结合能力的Cas蛋白突变体(dCas9/12)与各种效应因子融合来实现位点特异性基因调控[125]。由dCas介导的转录调控不会引起双链或单链的断裂,从而避免对宿主细胞的伤害,在转录调控方面具有巨大的应用前景。目前,已与dCas9/12融合并应用于丝状真菌的调控因子主要包括转录激活因子、抑制因子和表观遗传修饰因子(图3),具体信息综述如下。
碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage 1 2016 ... 碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ Base editing: advances and therapeutic opportunities 0 2020 Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C: G-to-T: a base editors with higher efficiency and product purity 1 2017 ... 碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ 碱基编辑技术及其在微生物合成生物学中的应用 1 2023 ... 碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ Base editing technology and its application in microbial synthetic biology 1 2023 ... 碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ CRISPR/Cas9 genome editing to demonstrate the contribution of Cyp51A Gly138Ser to azole resistance in Aspergillus fumigatus 1 2018 ... 碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ Genome-wide target specificities of CRISPR RNA-guided programmable deaminases 1 2017 ... 碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ CRISPR-Cas9 base editors and their current role in human therapeutics 1 2023 ... 碱基编辑器是在CRISPR/Cas系统基础上,将突变型Cas与脱氨酶等其他功能蛋白融合,实现不同碱基的转换,最初是由David Liu团队在2016年 开发[ Development of an efficient C-to-T base-editing system and its application to cellulase transcription factor precise engineering in thermophilic fungus Myceliophthora thermophila 1 2022 ... 胞嘧啶碱基编辑器是将nCas9(nickase Cas9)与胞嘧啶脱氨酶和尿嘧啶糖基化酶抑制子融合,gRNA引导融合蛋白到靶位点时,暴露出来的非靶向DNA单链上的胞嘧啶C可被脱氨酶转换为尿嘧啶U,而U与胸腺嘧啶T的具有相同的碱基配对规则,在DNA修复时,尿嘧啶糖基化酶抑制子会抑制U→C的修复,提高U转换为T的修复效率,从而完成C→T的碱基转换[ Multiplex base-editing enables combinatorial epigenetic regulation for genome mining of fungal natural products 1 2023 ... 胞嘧啶碱基编辑器是将nCas9(nickase Cas9)与胞嘧啶脱氨酶和尿嘧啶糖基化酶抑制子融合,gRNA引导融合蛋白到靶位点时,暴露出来的非靶向DNA单链上的胞嘧啶C可被脱氨酶转换为尿嘧啶U,而U与胸腺嘧啶T的具有相同的碱基配对规则,在DNA修复时,尿嘧啶糖基化酶抑制子会抑制U→C的修复,提高U转换为T的修复效率,从而完成C→T的碱基转换[ CRISPR technology: a decade of genome editing is only the beginning 1 2023 ... CRISPR/Cas除了可用上述的基因编辑功能来灭活各种调控因子或替换启动子进而影响基因的表达,还可将丧失切割活性但仍具有RNA引导下与DNA结合能力的Cas蛋白突变体(dCas9/12)与各种效应因子融合来实现位点特异性基因调控[ Modular synthetic biology toolkit for filamentous fungi 1 2021 ... CRISPR介导的转录激活(CRISPRa)主要是通过将核酸内切酶失活的dCas9/12与转录激活因子融合,并在gRNA指导下结合到目标基因的启动子区域,从而增强特定基因的表达[ Highly efficient Cas9-mediated transcriptional programming 2 2015 ... CRISPR介导的转录激活(CRISPRa)主要是通过将核酸内切酶失活的dCas9/12与转录激活因子融合,并在gRNA指导下结合到目标基因的启动子区域,从而增强特定基因的表达[
Other Images/Table from this Article
|