|
|||||||||||||||||||||
|
Halogenases in Biocatalysis: Advances in Mechanism Elucidation, Directed Evolution, and Green Manufacturing
Synthetic Biology Journal
DOI: 10.12211/2096-8280.2024-091
Table 2
The uniqueness and superiority of the CLEAN algorithm framework
Extracts from the Article
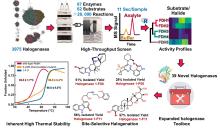
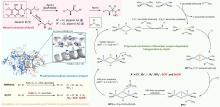
随着DNA测序技术的发展,特别是基因组学和宏基因组学工具的发展,人们发现了大量的蛋白质序列。然而,目前科学家只对蛋白质序列总量的0.3% (约50万)进行过研究,其中有明确实验证据支持的序列占比不足20%(<19.4%)[48-50]。另一方面,蛋白质功能注释高度依赖于计算模型的运行方式。一项大规模的蛋白质功能注释关键评估(CAFA)研究发现,使用现有计算工具自动注释的酶中有40%是不正确的[50]。因此,从有限的实验数据基础上对未知或未表征酶进行准确功能注释预测,对于蛋白质序列数据分析处理和应用至关重要。为了突破已有方法的局限性(表2),赵惠民团队开发了一种基于对比学习的机器学习模型CLEAN(Contrastive Learning-enabled Enzyme ANnotation)[51]。在CLEAN的任务中,具有相同酶分类编号(EC,Enzyme commission,指示酶催化何种反应的ID代码)的氨基酸序列具有较小的欧氏距离,反映了功能相似性。CLEAN基于UniProt的高质量数据库进行训练,输入氨基酸序列,随后计算对比输入样本间的欧式距离,就可得到EC可能性排序列表。为了验证CLEAN的准确性和稳健性,针对UniProt数据库中样本有限,研究不足的卤化酶数据集,使用CLEAN及其他六种最先进的注释工具(ProteInfer、BLASTp、DeepEC、DEEPre、COFACTOR和ECPred)对36种未完全注释的卤化酶的EC编号进行重新分配,结合实验验证结果,综合表明CLEAN(预测精度86.7至100%)在酶功能预测任务中比其他工具(例如,DeepEC中约11.1%,ProteInfer中11.1至61.1%)更具优越性。此外,所有预测工具中只有CLEAN成功将原本功能注释不准确甚至相互矛盾的卤化酶MJ1651(EC 3.13.1.8)和TTHA0338(EC 3.13.1.8)精准区分,并准确识别SsFlA多功能酶具备三种催化能力(EC 2.5.1.63, EC 2.5.1.94和EC 3.13.1.8)。结果表明,CLEAN能够高效处理低相似性序列、数据不平衡及混杂酶等复杂数据集,预测准确率显著提升并兼具抗噪性和纠错能力,为酶功能注释提供高精度、强鲁棒的计算工具。此外,该项工作证实了机器学习等AI技术在卤化酶识别、分类、鉴定方面的高效性和准确性,具有巨大应用潜力。
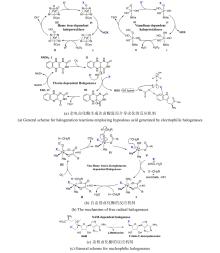
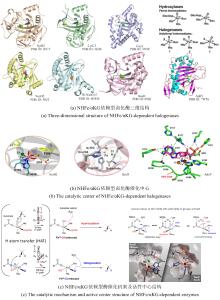
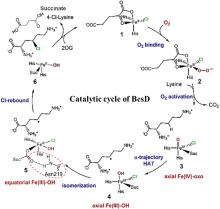
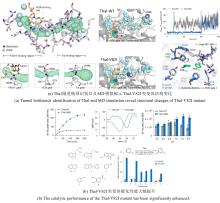
Kulik及其研究小组的计算工作详细描述了底物进入SyrB2活性位点所需的蛋白质间相互作用[79]。当苏氨酸被非天然氨基酸底物(如α-氨基丁酸或正缬氨酸)替代时,SyrB2主要催化羟化反应。这些研究表明,SyrB2与苏氨酸及其他氨基酸底物的相互作用对卤化/羟化反应的结果具有重要影响。尽管对SyrB2中的底物-蛋白相互作用进行了研究,但尚不清楚Fe中心SCS,即与Fe中心各配体相互作用的氨基酸残基)如何影响SyrB2和其他卤化酶的催化结果。Wilson等[68]通过生信分析发现,与SyrB2同源性较高的卤化酶中121位氨基酸残基为高度保守的苯丙氨酸,而羟化酶中则多为氧化还原性质活泼的酪氨酸。随后,将SyrB2催化中心SCS中的苯丙氨酸替换为酪氨酸(F121Y和F104Y),作为监测活性中心Cl-Fe配位方式的空间约束探针。通过分子动力学模拟以及实验证明,仅有F121Y突变体的酪氨酸在催化过程中与Fe(IV)=O反应形成DOPA,并导致SyrB2卤化功能丧失,而F104Y则没有。根据两突变体SCS中酪氨酸残基与Fe(IV)=O各配体之间的距离计算结果,确认了SyrB2卤化过程中Fe(IV)=O唯一的活性构象,即轴向Fe(IV)-Cl。
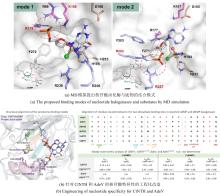
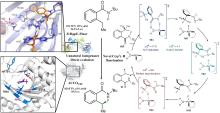
红线表示对氟化活性至关重要的残基;黄线表示能与腺嘌呤C2′位点修饰基团形成氢键的残基
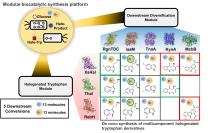
近期,唐奕课题组从Penicillium oxalicum中鉴定并表征了自由基卤化酶家族中的首个铜依赖型成员ApnU[ Regioselective halogenation of lavanducyanin by a site-selective vanadium-dependent chloroperoxidase 1 2024 ... 此外,非血红素铁/α-酮戊二酸依赖型卤化酶家族也增添了新成员AdeV和CtNTH,分别催化2'-脱氧腺苷-5'-单磷酸(dAMP)和2'-脱氧鸟苷-5'-单磷酸(dGMP)的氯化[ UniProt: the universal protein knowledgebase in 2021 1 2021 ... 随着DNA测序技术的发展,特别是基因组学和宏基因组学工具的发展,人们发现了大量的蛋白质序列.然而,目前科学家只对蛋白质序列总量的0.3% (约50万)进行过研究,其中有明确实验证据支持的序列占比不足20%(<19.4%)[ An unusual aromatase/cyclase programs the formation of the phenyldimethylanthrone framework in anthrabenzoxocinones and fasamycin 0 2024 A large-scale evaluation of computational protein function prediction 2 2013 ... 随着DNA测序技术的发展,特别是基因组学和宏基因组学工具的发展,人们发现了大量的蛋白质序列.然而,目前科学家只对蛋白质序列总量的0.3% (约50万)进行过研究,其中有明确实验证据支持的序列占比不足20%(<19.4%)[
红线表示对氟化活性至关重要的残基;黄线表示能与腺嘌呤C2′位点修饰基团形成氢键的残基 ...
红线表示对氟化活性至关重要的残基;黄线表示能与腺嘌呤C2′位点修饰基团形成氢键的残基 ...
Other Images/Table from this Article
|